Arkea
| Arkea Rentang waktu:
| |
|---|---|
| |
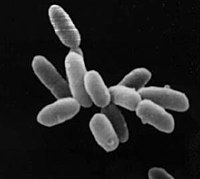
| Halobacteria sp. strain NRC-1, setiap sel memiliki panjang sekitar 5 μm | |
| Klasifikasi ilmiah | |
| Domain: | Archaea |
| Kerajaan[4] dan filum[5] | |
| |
| Sinonim | |
| |
Arkea atau Archaea merupakan domain mikroorganisme bersel satu. Arkea tergolong prokariota, kelompok mikrob yang tidak memiliki inti sel atau organel yang dibatasi membran. Awalnya, arkea diklasifikasikan sebagai bakteri dan diberi nama archaebacteria (di kerajaan Archaebacteria), tetapi klasifikasi ini tak lagi digunakan.
Sel arkea memiliki sifat unik yang memisahkan arkea dari dua domain kehidupan yang lain, yaitu Bacteria dan Eukaryota. Arkea dibagi lagi menjadi beberapa filum. Klasifikasinya sulit dilakukan karena mayoritas arkea belum diisolasi di laboratorium dan hanya dideteksi urutan gennya dari sampel yang diambil dari lingkungan.
Arkea dan bakteri umumnya memiliki ukuran dan bentuk yang serupa, meskipun beberapa arkea memiliki bentuk yang sangat aneh, misalnya sel-sel datar dan berbentuk persegi seperti Haloquadratum walsbyi. Meskipun terdapat kesamaan morfologi antara arkea dengan bakteri, arkea memiliki gen-gen dan beberapa jalur metabolik yang lebih serupa dengan eukariota, terutama enzim yang terlibat dalam transkripsi dan translasi. Salah satu aspek biokimia arkea yang unik adalah ketergantungan arkea pada lipid eter di membran sel mereka. Arkea menggunakan lebih banyak sumber energi dibandingkan eukariota, yang berkisar dari senyawa organik seperti gula, amonia, ion logam, atau bahkan gas hidrogen. Arkea yang toleran terhadap garam (Haloarchaea) menggunakan sinar matahari sebagai sumber energi, sementara spesies arkea lain memfiksasi karbon; namun, tidak seperti tumbuhan dan sianobakteri, tidak ada arkea yang melakukan semua cara tersebut. Arkea bereproduksi secara aseksual dengan pembelahan biner, fragmentasi, atau tunas; tidak seperti bakteri dan eukariota, tidak ada spesies arkea yang diketahui membentuk endospora. Arkea pertama yang diamati merupakan ekstremofil yang hidup di lingkungan ekstrem, seperti mata air panas dan danau garam tanpa organisme lain. Meningkatnya kemampuan alat deteksi molekuler memungkinkan penemuan arkea di hampir semua habitat, termasuk tanah, lautan, dan rawa. Arkea sangat banyak di lautan, dan arkea dalam bentuk plankton mungkin merupakan salah satu kelompok organisme yang paling melimpah di planet ini.
Arkea berperan besar dalam kehidupan di Bumi. Mereka merupakan bagian dari mikrobiota bagi semua organisme. Sebagai mikrobiota manusia, arkea berperan penting di usus besar, rongga mulut, dan kulit. Keragaman morfologis, metabolik, dan geografis memungkinkan mereka memiliki peran ekologis yang luas, misalnya pada fiksasi karbon dalam siklus karbon, peran dalam siklus nitrogen, perputaran senyawa organik, dan memelihara komunitas simbiosis dan sintrofi mikrob. Belum ditemukan contoh yang jelas untuk arkea yang bersifat patogenik atau parasitik. Sebaliknya, arkea sering kali bersifat mutualis atau komensal, misalnya metanogen yang hidup di saluran pencernaan manusia dan ruminansia, tempat mereka hidup dalam jumlah besar dan membantu pencernaan. Metanogen digunakan dalam produksi biogas dan pengolahan limbah. Sementara itu, enzim dari arkea ekstremofil yang dapat bertahan pada suhu tinggi dan pelarut organik dimanfaatkan dalam bioteknologi.
Klasifikasi
Konsep awal
Pada hampir sepanjang abad ke-20, prokariota dianggap sebagai satu kelompok organisme dan diklasifikasikan berdasarkan biokimia, morfologi dan metabolisme mereka. Misalnya, mikrobiolog mencoba mengklasifikasikan mikroorganisme berdasarkan struktur dinding sel mereka, bentuk mereka, dan zat-zat yang mereka konsumsi.[6] Pada tahun 1965, Linus Pauling dan Emile Zuckerkandl[7] mengusulkan untuk menggunakan urutan gen dalam berbagai prokariota untuk mengetahui bagaimana mereka berhubungan satu sama lain. Pendekatan ini, yang dikenal sebagai filogenetik, adalah metode utama yang digunakan saat ini.[8]
Arkea–yang diketahui pada saat itu hanya metanogen–pertama kali diklasifikasikan sebagai kelompok yang terpisah dari bakteri pada tahun 1977 oleh Carl Woese dan George E. Fox pada pohon filogenetik berdasarkan urutan gen RNA ribosomal (rRNA) mereka.[9] Kedua kelompok tersebut awalnya diberi nama Archaebacteria dan Eubacteria dan diperlakukan sebagai kerajaan atau subkerajaan, yang oleh Woese dan Fox disebut Urkingdoms. Namun, klasifikasi ini tak lagi digunakan.[10] Woese lalu berpendapat bahwa kelompok ini merupakan semacam prokariota yang secara fundamental berbeda dari kehidupan yang lain: (1) ketiadaan peptidoglikan pada dinding sel mereka; (2) adanya dua koenzim yang tidak biasa; dan (3) hasil pengurutan gen RNA ribosomal 16S. Untuk menekankan perbedaan ini, Woese kemudian mengusulkan sistem pengelompokan organisme yang baru dengan tiga domain terpisah: Eukarya, Bacteria dan Archaea,[1] yang sekarang dikenal sebagai "Revolusi Woesian".[11]
Kata arkea berasal dari bahasa Yunani Kuno ἀρχαῖα, yang berarti "hal-hal kuno",[12] karena wakil pertama dari domain Archaea adalah metanogen dan diasumsikan bahwa metabolisme mereka mencerminkan suasana primitif bumi dan organisme kuno. Untuk waktu yang lama, arkea terlihat sebagai ekstremofil yang hanya ada di habitat ekstrem seperti mata air panas dan danau garam. Namun, seiring bertambahnya habitat baru yang dipelajari, organisme yang lebih banyak juga ditemukan. Halofilik ekstrem[13] dan hipertermofilik mikrob[14] juga termasuk dalam Archaea. Pada akhir abad ke-20, arkea juga telah diidentifikasi dalam lingkungan non-ekstrem. Saat ini, mereka dikenal sebagai kelompok organisme yang besar dan beragam yang tersebar luas di alam.[15] Apresiasi baru tentang pentingnya dan keberadaan arkea berasal dari penggunaan reaksi berantai polimerase (PCR) untuk mendeteksi prokariota dari sampel lingkungan (seperti air atau tanah) dengan melipatgandakan gen ribosom mereka. Hal ini memungkinkan deteksi dan identifikasi organisme yang belum dibudidayakan di laboratorium.[16][17]
Klasifikasi
Klasifikasi arkea, dan prokariota pada umumnya, adalah bidang yang bergerak cepat dan diperdebatkan. Sistem klasifikasi saat ini bertujuan untuk mengatur arkea ke dalam kelompok organisme yang berbagi karakteristik struktural dan nenek moyang yang sama.[18] Klasifikasi ini sangat bergantung pada penggunaan urutan gen RNA ribosomal untuk mengungkapkan hubungan di antara berbagai organisme (filogenetik molekuler).[19] Sebagian besar spesies yang dapat dikultur dan diteliti dengan baik dari arkea adalah anggota dari dua filum utama, Euryarchaeota dan Crenarchaeota. Kelompok-kelompok lain telah dibuat secara tentatif. Misalnya, spesies aneh Nanoarchaeum equitans, yang ditemukan pada tahun 2003, telah diberikan filum sendiri, Nanoarchaeota.[20] Sebuah filum baru Korarchaeota juga telah diusulkan. Filum ini berisi sekelompok kecil spesies termofilik biasa yang berbagi karakteristik dari kedua filum utama, tetapi yang paling erat terkait dengan Crenarchaeota.[21][22] Spesies arkea lain yang baru terdeteksi ternyata terkait jauh dengan salah satu dari kelompok ini, seperti Archaeal Richmond Mine acidophilic nanoorganisms (ARMAN) yang ditemukan pada tahun 2006[23] dan merupakan salah satu organisme terkecil yang diketahui.[24]
Sebuah superfilum, yang disebut TACK, telah diusulkan yang mencakup Thaumarchaeota, Aigarchaeota, Crenarchaeota, dan Korarchaeota.[25] Superfilum ini mungkin terkait dengan asal usul eukariota. Pada tahun 2017, superfilum Asgard telah diajukan sebagai kelompok yang lebih dekat dengan eukariota asli dan merupakan kelompok saudari dari TACK.[26]
Kladogram
Berikut ini adalah kladogram arkea berdasarkan Tom A. Williams dkk. (2017)[27] serta Castelle dan Banfield (2018)[28] (DPANN):
|
Archaea |
Konsep spesies
Klasifikasi spesies-spesies arkea juga kontroversial. Biologi mendefinisikan spesies sebagai sebuah kelompok dari organisme yang saling terkait. Kriteria perkembangbiakan eksklusif yang diketahui secara umum (organisme yang dapat berkembang biak dengan satu sama lain tetapi tidak dengan yang lain) tidak membantu di sini karena arkea bereproduksi secara aseksual.[30]
Arkea menunjukkan tingkat transfer gen horizontal yang tinggi di antara garis keturunan mereka. Beberapa peneliti menyarankan bahwa sejumlah individu dapat dikelompokkan menjadi populasi seperti-spesies dengan genom yang sangat mirip dan jarang mentransfer gen ke/dari sel dengan genom kurang terkait, seperti dalam genus Ferroplasma.[31] Di sisi lain, studi terhadap Halorubrum menemukan adanya transfer genetik yang signifikan ke/dari populasi yang kurang terkait, yang membatasi penerapan kriteria ini.[32] Kekhawatiran kedua adalah sejauh mana sebutan spesies memiliki arti praktis.[33]
Pengetahuan terkini tentang keanekaragaman genetik masih tidak lengkap dan jumlah spesies arkea tidak dapat diperkirakan dengan akurat.[19] Perkiraan jumlah filum berkisar antara 18–23, tetapi hanya delapan filum yang memiliki perwakilan yang telah dibudidayakan dan dipelajari secara langsung. Banyak dari kelompok-kelompok hipotesis dikenal dari urutan rRNA tunggal, yang menunjukkan bahwa perbedaan di antara organisme ini tetap tidak jelas.[34] Bakteri juga mengandung banyak mikrob yang belum dikultur dengan implikasi yang sama untuk karakterisasi.[35]
Asal-usul dan evolusi
Umur Bumi diperkirakan sekitar 4,54 miliar tahun.[36][37][38] Bukti ilmiah menunjukkan bahwa kehidupan di Bumi dimulai setidaknya 3,5 miliar tahun yang lalu.[39][40] Bukti-bukti awal untuk kehidupan di Bumi adalah grafit yang bersifat biogenik pada batuan metasedimentari berumur 3,7 miliar tahun yang ditemukan di Greenland Barat[41] dan fosil tikar mikrob yang ditemukan di batu pasir setua 3,48 miliar tahun yang ditemukan di Australia Barat.[42][43] Pada tahun 2015, "sisa-sisa kehidupan biotik" ditemukan di batuan berumur 4,1 miliar tahun di Australia Barat.[44][45]
Meskipun fosil sel prokariotik kemungkinan berasal dari hampir 3,5 miliar tahun yang lalu, sebagian besar prokariota tidak memiliki morfologi yang dapat dibedakan, dan bentuk fosil tidak dapat digunakan untuk mengidentifikasi mereka sebagai arkea.[46] Sebaliknya, fosil kimia lipid yang unik menjadi lebih informatif karena senyawa tersebut tidak ada pada organisme lain.[47] Beberapa publikasi menyatakan bahwa sisa-sisa lipid arkea atau eukariota yang hadir dalam serpih berasal dari 2,7 miliar tahun yang lalu;[48] meskipun data tersebut dipertanyakan.[49] Lipid tersebut juga telah terdeteksi di batuan yang bahkan lebih tua dari Greenland barat. Jejak tertua berasal dari distrik Isua, Greenland, yang termasuk sedimen tertua yang dikenal di Bumi yang terbentuk 3,8 miliar tahun yang lalu.[50] Silsilah arkea mungkin menjadi garis keturunan yang paling kuno yang ada di Bumi.[51]
Woese berpendapat bahwa bakteri, arkea, dan eukariota merupakan garis keturunan yang terpisah yang menyimpang sejak awal dari koloni leluhur organisme.[52][53] Salah satu kemungkinannya[53][54] adalah bahwa percabangan ini terjadi sebelum evolusi sel, saat tidak adanya membran sel memperbolehkan transfer gen lateral yang tak terbatas, dan bahwa nenek moyang dari tiga domain muncul dengan fiksasi subset spesifik gen.[53][54] Ada kemungkinan bahwa nenek moyang terakhir dari bakteri dan arkea adalah termofil, yang meningkatkan kemungkinan bahwa suhu yang lebih rendah merupakan "lingkungan yang ekstrem" bagi arkea, dan organisme yang hidup di lingkungan dingin baru muncul hanya setelahnya.[55] Karena Archaea dan Bacteria tidak lebih terkait satu sama lain dibandingkan antara mereka dengan eukariota, istilah prokariota mungkin menunjukkan kesamaan yang salah di antara mereka.[56]
Perbandingan dengan domain lain
Tabel berikut membandingkan beberapa karakteristik utama dari tiga domain, untuk menggambarkan persamaan dan perbedaan mereka.[57]
| Properti | Archaea | Bacteria | Eukarya |
|---|---|---|---|
| Membran sel | Lipid dengan ikatan eter | Lipid dengan ikatan ester | Lipid dengan ikatan ester |
| Dinding sel | Pseudopeptidoglikan, glikoprotein, atau lapisan-S | Peptidoglikan, lapisan-S, atau tanpa dinding sel | Beragam struktur |
| Struktur gen | Kromosom melingkar, translasi dan transkripsi hampir sama dengan Eukarya | Kromosom melingkar, translasi and transkripsi unik | Kromosom linear yang jumlahnya lebih dari satu, translasi dan transkripsi hampir sama dengan Archaea |
| Struktur internal sel | Organel (?[58]) atau nukleus tidak dibatasi membran | Organel atau nukleus tidak dibatasi membran | Organel dan nukleus dibatasi membran |
| Metabolisme[59] | Bermacam-macam, termasuk diazotropi (pengikatan nitrogen), dengan metanogenesis unik untuk Archaea | Bermacam-macam, termasuk fotosintesis, respirasi aerobik dan anaerobik, fermentasi, diazotrofi, dan autotrofi | Fotosintesis, respirasi seluler, fermentasi; tidak ada diazotrofi |
| Reproduksi | Reproduksi aseksual, transfer gen horizontal | Reproduksi aseksual, transfer gen horizontal | Reproduksi seksual dan aseksual |
| Inisiasi sintesis protein | Metionin | Formilmetionin | Metionin |
| Polimerase RNA | Banyak | Satu | Banyak |
| Toksin | Sensitif terhadap toksin difteri | Resistan terhadap toksin difteri | Sensitif terhadap toksin difteri |
Archaea dipisahkan sebagai domain ketiga karena perbedaan besar dalam struktur RNA ribosomal mereka. Molekul RNA tertentu yang dikenal sebagai RNA ribosomal 16S (biasanya disingkat menjadi 16s rRNA) merupakan kunci penting dalam produksi protein di semua organisme. Karena fungsi ini jadi pusat kehidupan, organisme yang mengalami mutasi 16s rRNA tidak mungkin bertahan hidup sehingga struktur nukleotida ini menjadi sangat stabil (tetapi tidak absolut) selama beberapa generasi. Ukuran 16s rRNA juga cukup besar untuk menyimpan informasi spesifik pada organisme tertentu, tetapi cukup kecil untuk diurutkan dan dibandingkan dalam waktu singkat. Pada tahun 1977, Carl Woese, seorang ahli mikrobiologi yang mempelajari urutan genetik organisme, mengembangkan metode perbandingan baru yang melibatkan pemecahan RNA menjadi fragmen yang bisa diurutkan dan dibandingkan dengan fragmen lain dari organisme lain.[9] Semakin mirip pola fragmen tersebut di antara spesies-spesies, hubungan kekerabatannya menjadi lebih dekat.[60]
Woese menggunakan metode perbandingan rRNA barunya untuk mengelompokkan dan mengkontraskan organisme yang berbeda. Ia mengurutkan berbagai spesies yang berbeda dan spesies-spesies ini ternyata berada pada kelompok metanogen dengan pola yang sangat berbeda dibandingkan prokariota atau eukariota yang telah dikenal.[9] Metanogen ini jauh lebih mirip satu sama lain dibandingkan dengan organisme lain yang diurutkan, yang mengarahkan Woese untuk mengusulkan domain baru Archaea.[9] Eksperimen Woese menunjukkan bahwa arkea lebih mirip dengan eukariota alih-alih prokariota, meskipun struktur mereka lebih mirip dengan prokariota.[61] Hal ini menyimpulkan bahwa Archaea dan Eukarya berbagi nenek moyang bersama yang lebih baru dibandingkan dengan Eukarya dan Bacteria.[61] Perkembangan inti sel terjadi setelah percabangan antara bakteri dan nenek moyang bersama ini.[61]
Salah satu sifat arkea yang unik adalah penggunaan lipid dengan ikatan eter secara berlimpah di membran sel mereka. Ikatan eter lebih stabil secara kimiawi dari ikatan ester yang ditemukan dalam Bacteria dan Eukarya, yang mungkin menjadi faktor yang berkontribusi terhadap kemampuan banyak arkea untuk bertahan di lingkungan ekstrem yang menempatkan stres berat pada membran sel, seperti panas ekstrem dan salinitas. Analisis komparatif genom arkea juga telah mengidentifikasi beberapa indel khusus yang terkonservasi secara molekuler dan protein tertentu yang secara unik ditemukan baik di semua arkea maupun di beberapa kelompok utama yang berbeda dalam arkea .[62][63][64] Sifat lain yang unik dari arkea adalah bahwa tidak ada organisme lain yang dikenal mampu melakukan metanogenesis (produksi metabolis metana). Arkea metanogen memainkan peran penting dalam ekosistem dengan organisme yang memperoleh energi dari oksidasi metana (kebanyakan dari mereka adalah bakteri) karena mereka sering menjadi sumber utama metana di lingkungan tersebut dan dapat berperan sebagai produsen primer. Metanogen juga memainkan peran penting dalam siklus karbon, memecah karbon organik menjadi metana, yang juga merupakan gas rumah kaca utama.[65]
Hubungan dengan bakteri
Hubungan antara tiga domain sangat penting untuk memahami asal-usul kehidupan. Sebagian besar lintasan metabolisme, yang merupakan objek dari sebagian besar gen suatu organisme, adalah hal yang umum ditemukan di antara Archaea dan Bacteria, sementara sebagian besar gen yang terlibat dalam ekspresi genom umum ditemukan di antara Archaea dan Eukarya.[67] Di antara prokariota, struktur sel arkea paling mirip dengan bakteri Gram-positif, terutama karena keduanya memiliki lipida dwilapis tunggal[68] dan biasanya mengandung sakulus tebal (eksoskeleton) dari berbagai komposisi kimia.[69] Pada beberapa pohon filogenetik yang didasarkan pada urutan gen/protein yang berbeda dari homolog prokariotik, homolog arkea lebih berhubungan dekat dengan homolog bakteri Gram-positif.[68] Arkea dan bakteri Gram-positif juga berbagi indel yang dilestarikan dalam sejumlah protein penting, seperti Hsp70 dan glutamin sintetase I;[68][70] namun, filogeni dari gen-gen ini ditafsirkan untuk mengungkapkan transfer gen interdomain,[71][72] dan mungkin tidak mencerminkan hubungan organisme.[73]
Ada pendapat bahwa arkea berevolusi dari bakteri Gram-positif sebagai respons terhadap tekanan seleksi antibiotik.[68][70][74] Pandangan ini diajukan berdasarkan pengamatan bahwa arkea tahan terhadap berbagai antibiotik yang terutama dibuat untuk bakteri Gram-positif,[68][70] dan bahwa antibiotik-antibiotik ini terutama bekerja pada gen yang membedakan arkea dari bakteri. Pendapat bahwa tekanan selektif terhadap resistansi yang dihasilkan oleh antibiotik Gram-positif akhirnya cukup untuk menyebabkan perubahan yang luas pada banyak gen target antibiotik, dan bahwa galur ini mewakili nenek moyang arkea masa kini.[74] Evolusi arkea dalam menanggapi pemilihan antibiotik, atau tekanan selektif kompetitif lain, juga bisa menjelaskan adaptasi mereka terhadap lingkungan yang ekstrem (seperti suhu tinggi atau keasaman) sebagai hasil dari pencarian relung kosong untuk melarikan diri dari organisme yang memproduksi antibiotik;[74][75] Cavalier-Smith juga membuat usulan yang sama.[76] Pendapat ini juga didukung oleh studi lainnya yang menyelidiki hubungan protein struktural[77] dan studi yang menunjukkan bahwa bakteri Gram-positif mungkin merupakan awal garis keturunan yang bercabang paling awal dalam prokariota.[78]
Hubungan dengan eukariota
Hubungan evolusi antara arkea dan eukariota masih belum jelas. Selain dari kesamaan dalam struktur dan fungsi sel yang dibahas di bawah, banyak pohon genetik yang mengelompokkan keduanya.[79]
Faktor yang memperrumit termasuk klaim bahwa hubungan antara eukariota dan filum arkea Crenarchaeota lebih dekat daripada hubungan antara Euryarchaeota dan filum Crenarchaeota[80] dan adanya gen seperti-arkea pada bakteri tertentu, seperti Thermotoga maritima, dari transfer gen horizontal.[81] Hipotesis standar menyatakan bahwa nenek moyang eukariota berpisah dari Archaea sejak awal,[82][83] dan bahwa eukariota muncul melalui fusi dari arkea dan bakteri, yang menjadi inti sel dan sitoplasma; hal ini menjelaskan berbagai kesamaan genetik tetapi kesulitan dalam menjelaskan struktur sel.[80] Sebuah hipotesis alternatif, hipotesis eosit, berpendapat bahwa Eukaryota muncul relatif terlambat dari Archaea.[84]
Sebuah garis keturunan arkea yang ditemukan pada 2015, Lokiarchaeum (dari filum baru Lokiarchaeota), yang dinamai berdasarkan sebuah ventilasi hidrotermal yang disebut Kastil Loki di Samudra Arktik, telah ditemukan sebagai kelompok yang paling berkerabat dengan eukariota. Organisme ini telah disebut organisme peralihan antara prokariota dan eukariota.[85][86]
Beberapa filum saudara dari "Lokiarchaeota" telah ditemukan ("Thorarchaeota", "Odinarchaeota", "Heimdallarchaeota"), yang semuanya membentuk sebuah supergrup Asgard yang baru diajukan, yang mungkin merupakan takson saudara dari Proteoarchaeota.[5][26][87] Rincian dari hubungan anggota Asgard dan eukariota masih dalam pertimbangan,[88] meskipun, pada Januari 2020, para ilmuwan melaporkan bahwa Candidatus Prometheoarchaeum syntrophicum, sejenis arkea Asgard, mungkin merupakan penghubung antara mikroorganisme prokariotik yang sederhana dan mikroorganisme eukariotik yang kompleks sekitar dua miliar tahun yang lalu.[89][90]
Morfologi
Satu individu arkea memiliki diameter yang berkisar dari 0,1 mikrometer (μm) sampai lebih dari 15 μm, yang hadir dalam berbagai bentuk, umumnya sebagai bola, batang, spiral atau piring.[91] Morfologi lain pada filum Crenarchaeota, termasuk sel berlobus yang bentuknya tidak teratur pada genus Sulfolobus, filamen seperti-jarum yang diameternya kurang dari setengah mikrometer pada genus Thermofilum, dan batang yang bentuknya hampir persegi panjang sempurna pada Thermoproteus dan Pyrobaculum.[92] Haloquadratum walsbyi adalah arkea dengan sel berbentuk persegi yang datar, yang hidup di kolam hipersalin.[93][94] Bentuk yang tidak biasa ini mungkin dipertahankan dengan baik oleh dinding sel dan sitoskeleton prokariotik mereka. Protein yang terkait dengan komponen sitoskeleton organisme lain ditemukan di arkea,[95] dan filamen terbentuk dalam sel mereka,[96] tetapi berbeda dengan organisme lain, struktur seluler ini kurang dipahami.[97] Dalam Thermoplasma dan Ferroplasma, ketiadaan dinding sel berarti bahwa sel memiliki bentuk yang tidak teratur, dan dapat menyerupai ameba.[98]
Beberapa spesies membentuk agregat atau filamen sel yang panjangnya hingga 200 μm.[91] Organisme ini bisa menonjol dalam biofilm.[99] Khususnya, agregat sel Thermococcus coalescens menyatu bersama dalam kultur, membentuk sel-sel raksasa tunggal.[100] Genus Pyrodictium menghasilkan koloni multisel rumit yang melibatkan tabung berongga panjang dan tipis yang disebut kanula yang menonjol dari permukaan sel dan menghubungkan mereka ke dalam aglomerasi padat seperti-semak.[101] Fungsi dari kanula ini tidak disepakati, tetapi mereka dapat memungkinkan komunikasi atau pertukaran nutrisi dengan tetangganya.[102] Koloni multispesies juga ditemukan, seperti komunitas "untaian mutiara" yang ditemukan pada tahun 2001 di sebuah rawa di Jerman. Koloni bulat keputihan dari spesies baru Euryarchaeota terletak di sepanjang filamen tipis yang panjangnya dapat mencapai 15 sentimeter (5,9 inci); filamen ini terbuat dari spesies bakteri tertentu.[103]
Struktur, perkembangan komposisi, dan operasi
Arkea dan bakteri memiliki struktur sel yang umumnya sama, tetapi komposisi dan organisasi sel memisahkan arkea dari bakteri. Seperti bakteri, arkea tidak memiliki membran interior dan organel.[56] Membran sel arkea, juga seperti bakteri, biasanya dibatasi oleh dinding sel dan mereka berenang menggunakan satu flagela atau lebih.[104] Secara struktural, arkea paling mirip dengan bakteri Gram-positif. Kebanyakan arkea memiliki membran plasma dan dinding sel tunggal, dan tidak ada ruang periplasmik; pengecualian untuk aturan umum ini adalah Ignicoccus, yang memiliki sebuah periplasma sangat besar yang berisi vesikel bermembran dan tertutup oleh membran luar.[105]
Dinding sel dan flagela
Kebanyakan arkea (kecuali Thermoplasma dan Ferroplasma) memiliki dinding sel.[98] Mayoritas arkea dinding selnya dirakit dari protein lapisan permukaan, yang membentuk lapisan-S.[106] Lapisan-S adalah susunan molekul protein yang kaku yang menutupi bagian luar sel (seperti zirah rantai).[107] Lapisan ini menyediakan perlindungan kimia dan fisik, dan dapat mencegah makromolekul mengalami kontak dengan membran sel.[108] Tidak seperti bakteri, arkea tidak memiliki peptidoglikan di dinding sel mereka.[109] Methanobacteriales memiliki dinding sel yang mengandung pseudopeptidoglikan, yang menyerupai peptidoglikan eubacteria dalam morfologi, fungsi, dan struktur fisik, tetapi pseudopeptidoglikan berbeda dalam struktur kimia; tidak memiliki asam amino-D dan asam N-asetilmuramat.[108]
Flagela arkea beroperasi seperti flagela bakteri—tangkai panjangnya digerakkan oleh motor yang berputar di pangkalnya. Motor ini didukung oleh gradien proton yang melintasi membran. Namun, flagela arkea berbeda, terutama dalam komposisi dan pengembangan.[104] Kedua jenis flagela (milik bakteri dan arkea) berevolusi dari nenek moyang yang berbeda. Flagela bakteri memiliki satu nenek moyang dengan sistem sekresi tipe III,[110][111] sementara flagela arkea tampaknya berevolusi dari pili bakteri tipe IV.[112] Berbeda dengan flagela bakteri, yang berongga dan dirakit oleh subunit yang bergerak naik dari pori tengah ke ujung flagela, flagela arkea disintesis dengan menambahkan subunit di pangkalnya.[113]
Membran
Membran arkea terbuat dari molekul yang sangat berbeda dari molekul di bentuk kehidupan lain, yang menunjukkan bahwa arkea terkait jauh dari bakteri dan eukariota.[114] Pada semua organisme, membran sel terbuat dari molekul yang dikenal sebagai fosfolipid. Molekul-molekul ini memiliki bagian kutub yang larut dalam air ("kepala" fosfat) dan bagian non-polar "berminyak" yang tidak larut dalam air (ekor lipid). Bagian yang berbeda ini dihubungkan oleh gugus gliserol. Dalam air, fosfolipid mengelompok, dengan kepala menghadap air dan ekor menjauhi air. Struktur utama dalam membran sel adalah lapisan ganda fosfolipid ini, yang disebut lipida dwilapis.[115]
Fosfolipid yang dimiliki arkea tidak biasa dalam empat hal:
- Bakteri dan eukariota memiliki membran yang terutama tersusun atas gliserol-lipid ester, sedangkan arkea memiliki membran yang tersusun atas gliserol-lipid eter.[116][117] Perbedaannya adalah jenis ikatan yang menggabungkan lipid ke bagian gliserol; dua jenis ikatan ditunjukkan dengan warna kuning pada gambar di sebelah kanan. Pada lipid ester, ikatan ini merupakan ikatan ester, sedangkan pada lipid eter ikatan ini merupakan ikatan eter. Ikatan eter secara kimiawi lebih tahan dari ikatan ester. Stabilitas ini mungkin membantu arkea bertahan pada suhu ekstrem dan lingkungan yang sangat asam atau basa.[118]
- Stereokimia dari bagian gliserol arkea berkebalikan dari yang ditemukan pada organisme lain. Bagian gliserol dapat terjadi dalam dua bentuk yang merupakan bayangan cermin satu sama lain, yang disebut bentuk tangan kanan dan tangan kiri; dalam kimia hal ini disebut enantiomer. Seperti tangan kanan tidak cocok dengan mudah ke dalam sarung tangan kiri, fosfolipid tangan kanan umumnya tidak dapat digunakan atau dibuat oleh enzim disesuaikan untuk bentuk kiri. Hal ini menunjukkan bahwa arkea menggunakan enzim yang sama sekali berbeda untuk sintesis fosfolipid dibandingkan bakteri dan eukariota. Enzim seperti ini dikembangkan sangat awal dalam sejarah kehidupan, yang menunjukkan percabangan awal dari dua domain lainnya.[114]
- Ekor lipid arkea secara kimiawi berbeda dari organisme lain. Lipid arkea didasarkan pada rantai isoprenoid panjang dengan banyak cabang samping, dan terkadang dengan cincin siklopropana atau sikloheksana.[119] Hal ini kontras dengan asam lemak yang ditemukan dalam membran organisme lain, yang memiliki rantai lurus tanpa cabang atau cincin. Meskipun isoprenoid memainkan peran penting dalam biokimia banyak organisme, hanya arkea yang menggunakannya untuk membuat fosfolipid. Rantai ini bercabang dapat membantu mencegah kebocoran membran arkea pada suhu tinggi.[120]
- Pada beberapa arkea, lapisan ganda lipid digantikan oleh sebuah lapisan tunggal. Akibatnya, arkea menyatukan dua ekor molekul fosfolipid independen menjadi molekul tunggal dengan dua kepala polar (bolaampifil); fusi ini dapat membuat membran mereka lebih kaku dan lebih mampu menahan lingkungan yang keras.[121] Sebagai contoh, lipid dalam Ferroplasma memiliki jenis lapisan ini, yang diduga untuk membantu kelangsungan hidup organisme ini di habitatnya yang sangat asam.[122]
Metabolisme
Arkea menunjukkan berbagai macam reaksi kimia dalam metabolisme mereka dan menggunakan banyak sumber energi. Reaksi-reaksi ini diklasifikasikan ke dalam kelompok nutrisional, tergantung pada sumber-sumber energi dan karbon. Beberapa arkea memperoleh energi dari senyawa anorganik seperti belerang atau amoniak (mereka disebut litotrof). Ini termasuk nitrifier, metanogen dan oksidan metana anaerob.[123] Dalam reaksi ini, satu senyawa memberikan elektron kepada senyawa yang lain (dalam reaksi redoks), melepaskan energi untuk bahan bakar aktivitas sel. Satu senyawa bertindak sebagai donor elektron dan satu sebagai akseptor elektron. Energi yang dilepaskan menghasilkan adenosina trifosfat (ATP) melalui kemiosmosis, dalam proses dasar yang sama yang terjadi di mitokondria sel eukariotik.[124]
Kelompok lain dari arkea menggunakan sinar matahari sebagai sumber energi (mereka disebut fototrof). Namun, fotosintesis yang menghasilkan oksigen tidak terjadi dalam organisme-organisme ini.[124] Banyak jalur metabolik dasar dimiliki bersama oleh semua bentuk kehidupan; misalnya, arkea menggunakan bentuk modifikasi dari glikolisis (jalur Entner–Doudoroff) serta salah satu antara siklus asam sitrat lengkap atau parsial.[125] Kesamaan dengan organisme lain ini mungkin mencerminkan awal mula dalam sejarah kehidupan dan tingkat efisiensi yang tinggi.[126]
| Tipe nutrisional | Sumber energi | Sumber karbon | Contoh |
|---|---|---|---|
| Fototrof | Cahaya matahari | Senyawa organik | Halobacterium |
| Litotrof | Senyawa anorganik | Senyawa organik atau fiksasi karbon | Ferroglobus, Methanobacteria, atau Pyrolobus |
| Organotrof | Senyawa organik | Senyawa organik atau fiksasi karbon | Pyrococcus, Sulfolobus, atau Methanosarcinales |
Beberapa Euryarchaeota adalah metanogen yang hidup di lingkungan anaerobik, seperti rawa-rawa. Bentuk metabolisme ini berevolusi sejak awal, dan bahkan mungkin bahwa organisme yang pertama hidup bebas adalah metanogen.[127] Reaksi umum melibatkan penggunaan karbon dioksida sebagai akseptor elektron untuk mengoksidasi hidrogen. Metanogenesis melibatkan berbagai koenzim yang unik untuk arkea ini, seperti koenzim M dan metanofuran.[128] Senyawa organik lain seperti alkohol, asam asetat, atau asam format digunakan sebagai alternatif akseptor elektron oleh metanogen. Reaksi-reaksi ini sering terjadi pada arkea yang tinggal di usus. Asam asetat juga dipecah menjadi metana dan karbon dioksida secara langsung oleh arkea asetotrofik. Asetotrof adalah arkea dalam ordo Methanosarcinales dan merupakan bagian utama dari komunitas mikroorganisme yang menghasilkan biogas.[129]
Arkea lainnya menggunakan CO2 di atmosfer sebagai sumber karbon dalam proses yang disebut fiksasi karbon (mereka disebut autotrof). Proses ini melibatkan bentuk yang sangat termodifikasi dari siklus Calvin[131] atau jalur metabolik yang baru ditemukan disebut siklus 3-hidroksipropionat/4-hidroksibutirat.[132] Crenarchaeota juga menggunakan siklus Krebs terbalik, sementara euryarchaeota juga menggunakan jalur reduktif asetil-KoA.[133] Fiksasi karbon ini didukung oleh sumber energi anorganik. Tidak ada arkea yang diketahui melakukan fotosintesis.[134] Sumber energi arkea sangat beragam, dan berkisar dari oksidasi amonia oleh Nitrosopumilales[135][136] sampai oksidasi hidrogen sulfida atau unsur belerang oleh spesies Sulfolobus, yang menggunakan oksigen atau ion logam sebagai akseptor elektron.[124]
Arkea fototrofik menggunakan cahaya untuk menghasilkan energi kimia dalam bentuk ATP. Dalam Halobacteria, pompa ion yang diaktifkan-cahaya seperti bakteriorodopsin dan halorodopsin menghasilkan gradien ion dengan cara memompa ion keluar dari sel melintasi membran plasma. Energi yang tersimpan dalam gradien elektrokimia kemudian diubah menjadi ATP oleh ATP sintase.[91] Proses ini merupakan bentuk fotofosforilasi. Kemampuan pompa didorong-cahaya ini untuk menggerakkan ion melintasi membran bergantung pada perubahan yang didorong-cahaya dalam struktur kofaktor retinol yang ada di tengah protein.[137]
Genetika
Arkea biasanya memiliki kromosom melingkar tunggal,[138] dengan ukuran hingga 5.751.492 pasangan basa pada Methanosarcina acetivorans,[139] genom arkea terbesar yang diketahui. Sepersepuluh dari ukuran ini adalah genom kecil berupa 490.885 pasangan basa Nanoarchaeum equitans, genom arkea terkecil yang dikenal; genom ini diperkirakan hanya berisi 537 gen penyandi-protein.[140] Potongan independen kecil dari DNA, yang disebut plasmid, juga ditemukan dalam arkea. Plasmid dapat ditransfer antara sel dengan kontak fisik, dalam proses yang mungkin mirip dengan konjugasi bakteri.[141][142]
Arkea secara genetik berbeda dari bakteri dan eukariota; sampai dengan 15% dari protein yang disandi oleh setiap genomnya merupakan protein yang unik untuk domain Archaea, meskipun sebagian besar gen yang unik tidak memiliki fungsi yang diketahui.[144] Dari sisa protein unik yang fungsinya telah diidentifikasi, sebagian besar dimiliki Euryarchaea dan terlibat dalam metanogenesis. Protein yang sama-sama dimiliki arkea, bakteri, dan eukariota membentuk inti umum dari fungsi sel, yang berkaitan terutama untuk transkripsi, translasi, dan metabolisme nukleotida.[145] Sifat arkea lain yang khas adalah organisasi gen terkait fungsi—seperti enzim yang mengkatalisis langkah dalam jalur metabolik yang sama ke dalam operon baru, dan perbedaan yang besar dalam gen tRNA dan tRNA sintetase aminoasilnya.[145]
Transkripsi pada arkea lebih menyerupai transkripsi eukariotik dibandingkan transkripsi bakteri, dengan RNA polimerase arkea yang sangat dekat dengan ekuivalen pada eukariota;[138] sementara translasi arkea menunjukkan tanda-tanda ekuivalen baik pada bakteri maupun eukariota.[146] Meskipun arkea hanya memiliki satu jenis RNA polimerase, struktur dan fungsinya dalam transkripsi tampaknya dekat dengan RNA polimerase II eukariotik, dengan kumpulan protein yang sama (faktor transkripsi umum) mengarahkan pengikatan RNA polimerase ke promotor gen.[147] Namun, faktor transkripsi arkea lainnya lebih dekat dengan yang ditemukan pada bakteri.[148] Modifikasi setelah-transkripsi lebih sederhana dibandingkan eukariota karena sebagian besar gen arkea tidak memiliki intron, meskipun ada banyak intron pada gen RNA transfer dan RNA ribosomal mereka,[149] dan intron mungkin ada pada beberapa gen penyandi-protein.[150][151]
Transfer gen dan pertukaran genetik
Halobacterium volcanii, sebuah arkea halofilik ekstrem, membentuk jembatan sitoplasma antara sel-sel yang sepertinya akan digunakan untuk transfer DNA dari satu sel ke sel lain dalam kedua arah.[152] Ketika arkea hipertermofilik Sulfolobus solfataricus[153] dan Sulfolobus acidocaldarius[154] terpapar agen perusak DNA radiasi sinar ultraungu (UV) atau bleomisin atau mitomisin C, agregasi seluler spesies-spesifik akan diinduksi. Agregasi pada S. solfataricus tidak dapat diinduksi oleh stres fisik lainnya, seperti pH atau perubahan suhu,[153] yang menunjukkan bahwa agregasi diinduksi secara khusus oleh kerusakan DNA. Ajon dkk.[154] menunjukkan bahwa agregasi seluler yang diinduksi sinar UV memediasi pertukaran penanda kromosom dengan frekuensi tinggi pada S. acidocaldarius. Tingkat rekombinasi arkea yang diinduksi melebihi kultur yang tidak diinduksi hingga tiga lipat. Frols dkk.[153][155] dan Ajon dkk.[154] berhipotesis bahwa agregasi seluler meningkatkan transfer DNA spesifik spesies antara sel-sel Sulfolobus dalam rangka memberikan peningkatan perbaikan DNA yang rusak dengan cara rekombinasi homolog. Tanggapan ini mungkin merupakan bentuk primitif dari interaksi seksual yang mirip dengan sistem transformasi bakteri yang dipelajari dengan lebih baik yang juga terkait dengan transfer DNA spesifik-spesies di antara sel-sel yang mengarah pada perbaikan rekombinasi homolog dari kerusakan DNA.[156]
Virus arkea
Arkea adalah target dari sejumlah virus yang berbeda dari virus yang menyerang bakteri dan eukariota. Virus-virus ini telah dikelompokkan dalam 15–18 keluarga berbasis DNA sejauh ini, tetapi beberapa spesies tetap tidak terisolasi dan menunggu klasifikasi.[157][158][159] Keluarga-keluarga ini secara informal dapat dibagi menjadi dua kelompok: virus spesifik arkea dan kosmopolitan. Virus spesifik arkea hanya menargetkan spesies arkea dan saat ini mencakup 12 keluarga virus. Banyak struktur virus unik yang sebelumnya tidak teridentifikasi telah diamati dalam kelompok ini, termasuk virus berbentuk botol, spindel, gulungan, dan tetesan.[158] Meskipun siklus reproduksi dan mekanisme genom spesies virus spesifik arkea mungkin mirip dengan virus lain, mereka memiliki karakteristik unik yang secara khusus dikembangkan karena morfologi sel inang yang mereka infeksi.[157] Mekanisme pelepasan virus mereka berbeda dari fag lainnya. Bakteriofag umumnya menjalani jalur litik, jalur lisogenik, atau campuran keduanya (meskipun jarang).[160] Sebagian besar galur virus spesifik arkea mempertahankan hubungan yang stabil dan agak lisogenik dengan inangnya, yang tampak sebagai infeksi kronis. Hal ini melibatkan produksi dan pelepasan virion secara bertahap dan terus-menerus tanpa membunuh sel inang.[161] Prangishyili (2013) mencatat bahwa telah dihipotesiskan bahwa fag arkea berekor berasal dari bakteriofag yang mampu menginfeksi spesies Haloarchaea. Jika hipotesisnya benar, dapat disimpulkan bahwa virus DNA beruntai ganda lainnya yang menyusun sisa kelompok spesifik arkea adalah kelompok unik mereka sendiri dalam komunitas virus global. Krupovic dkk. (2018) menyatakan bahwa tingginya tingkat transfer gen horizontal, tingkat mutasi yang cepat pada genom virus, dan kurangnya urutan gen universal telah membuat para peneliti mempersepsikan jalur evolusi virus arkea sebagai jejaring. Kurangnya kesamaan antara penanda filogenetik dalam jejaring ini dan virosfer global, serta hubungan eksternal dengan elemen non-virus, mungkin menunjukkan bahwa beberapa spesies virus spesifik arkea berevolusi dari elemen genetik seluler non-virus (MGE).[158]
Virus-virus ini telah dipelajari secara rinci pada kelompok termofil, khususnya ordo Sulfolobales dan Thermoproteales.[162] Dua kelompok virus DNA untai tunggal yang menginfeksi arkea telah baru-baru ini diisolasi. Satu kelompok dicontohkan oleh virus pleomorfik Halorubrum 1 ("Pleolipoviridae") yang menginfeksi arkea halofilik[163] dan yang lainnya oleh Aeropyrum coil berbentuk virus ("Spiraviridae") yang menginfeksi inang hipertermofilik (pertumbuhan optimal pada 90–95 °C).[164] Khususnya, virus yang terakhir dilaporkan memiliki genom ssDNA yang terbesar hingga saat ini. Pertahanan terhadap virus ini mungkin melibatkan interferensi RNA dari urutan DNA repetitif yang terkait dengan gen dari virus.[165][166]
Reproduksi
Arkea bereproduksi secara aseksual dengan fisi biner atau berganda, fragmentasi, atau tunas; mitosis dan meiosis tidak terjadi, jadi jika spesies arkea hadir dalam lebih dari satu bentuk, semua memiliki materi genetik yang sama.[91] Pembelahan sel dikendalikan dalam siklus sel; setelah kromosom sel direplikasi dan dua kromosom anak terpisah, sel akan membelah.[167] Dalam genus Sulfolobus, siklus sel memiliki karakteristik yang mirip dengan sistem bakteri dan eukariotik. Kromosom bereplikasi dari beberapa titik-awal (origo replikasi) menggunakan DNA polimerase yang menyerupai enzim eukariotik yang setara.[168]
Pada Euryarchaea, protein pembelahan sel FtsZ, yang membentuk cincin kontraktor di sekitar sel, dan komponen septum yang dibangun di tengah sel, mirip dengan protein yang setara pada bakteri.[167] Pada cren-[169][170] dan thaumarchaea,[171] mesin pembelahan sel CDV memiliki peran serupa. Mesin ini terkait dengan mesin eukariotik ESCRT-III (yang sementara paling dikenal untuk perannya dalam pemilahan sel) yang juga terlihat memenuhi peran dalam pemisahan pada sel yang membelah, yang menunjukkan peran leluhur dalam pembelahan sel.[172]
Baik bakteri dan eukariota, tetapi tidak arkea, membentuk spora.[173] Beberapa spesies Haloarchaea menjalani peralihan fenotipik dan tumbuh sebagai beberapa jenis sel yang berbeda, termasuk struktur berdinding tebal yang tahan terhadap guncangan osmotik dan memungkinkan arkea untuk bertahan hidup dalam air pada konsentrasi garam rendah, tetapi ini bukanlah struktur reproduksi dan dapat membantu mereka mencapai habitat baru.[174]
Ekologi
Habitat
Arkea ada di berbagai habitat dan kini diakui sebagai bagian besar dalam ekosistem global,[15] yang dapat berkontribusi hingga 20% dari biomassa bumi.[175] Arkea yang pertama ditemukan adalah ekstremofili.[123] Memang, beberapa arkea bertahan pada suhu tinggi, sering di atas 100 °C (212 °F), seperti yang ditemukan di geyser, perokok hitam, dan sumur minyak. Habitat umum lainnya termasuk habitat yang sangat dingin dan air yang sangat asin, asam, atau alkali. Namun, arkea mencakup mesofil yang tumbuh dalam kondisi ringan, di tanah rawa, air limbah, samudra, saluran usus hewan, dan tanah.[15]
Arkea ekstremofil adalah anggota dari empat kelompok fisiologis utama. Kelompok-kelompok itu adalah halofili, termofili, alkalifili, dan asidofili.[176] Kelompok-kelompok ini tidak komprehensif atau spesifik terhadap filum, mereka juga tidak saling eksklusif, karena beberapa arkea digolongkan dalam beberapa kelompok. Meskipun demikian, mereka adalah titik awal yang berguna untuk klasifikasi.[177]
Halofili, termasuk genus Halobacterium, hidup di lingkungan yang sangat asin seperti danau garam dan melebihi rekan-rekan bakteri mereka dengan salinitas lebih besar dari 20–25%.[123] Termofili tumbuh terbaik pada suhu di atas 45 °C (113 °F), di tempat-tempat seperti mata air panas; arkea hipertermofilik tumbuh optimal pada suhu lebih besar dari 80 °C (176 °F).[178] Arkea Methanopyrus kandleri galur 116 bahkan dapat bereproduksi pada 122 °C (252 °F), suhu tertinggi yang tercatat dari organisme manapun.[179]
Arkea lain hidup di kondisi yang sangat asam atau basa.[176] Sebagai contoh, salah satu asidofil arkea paling ekstrem adalah Picrophilus torridus, yang tumbuh pada pH 0, yang setara dengan berkembang di 1,2 molar asam sulfat.[180]
Ketahanan pada lingkungan yang ekstrem ini telah membuat arkea menjadi fokus spekulasi tentang kemungkinan sifat kehidupan ekstraterestrial.[181] Beberapa habitat ekstremofil tidak berbeda dengan Mars,[182] yang mengarah ke sugesti bahwa mikrob yang layak dapat ditransfer di antara planet-planet di meteorit.[183]
Baru-baru ini, beberapa penelitian telah menunjukkan bahwa arkea ada tidak hanya ada di lingkungan mesofilik dan termofilik tetapi juga hadir, kadang-kadang dalam jumlah yang tinggi, pada suhu rendah juga. Misalnya, arkea umum dalam lingkungan lautan dingin seperti lautan kutub.[184] Lebih signifikan lagi adalah temuan sejumlah besar arkea di seluruh lautan di dunia pada habitat non-ekstrem di antara komunitas plankton (sebagai bagian dari pikoplankton).[185] Meskipun arkea ini dapat hadir dalam jumlah yang sangat tinggi (hingga 40% dari biomassa mikrob), hampir tidak ada spesies ini yang telah diisolasi dan dipelajari dalam kultur murni.[186] Akibatnya, pemahaman kita tentang peran arkea di ekologi laut masih mendasar, sehingga pengaruh penuh mereka pada siklus biogeokimia global sebagian besar masih belum diselidiki.[187] Beberapa Crenarchaeota laut mampu melakukan nitrifikasi, yang menunjukkan bahwa organisme ini dapat mempengaruhi siklus nitrogen samudra,[135] meskipun Crenarchaeota laut ini juga dapat menggunakan sumber-sumber energi lain.[188]
Sejumlah besar arkea juga ditemukan dalam sedimen yang menutupi dasar laut, dengan organisme ini membentuk sebagian besar sel hidup pada kedalaman lebih dari 1 meter di bawah dasar laut.[189][190] Telah dibuktikan bahwa di semua sedimen permukaan samudera (dari kedalaman air 1.000 sampai 10.000 m), dampak infeksi virus lebih tinggi pada arkea dibandingkan pada bakteri; sementara itu, lisis arkea yang diinduksi virus menyumbang hingga sepertiga dari total biomassa mikrob yang terbunuh, yang menghasilkan pelepasan ~ 0,3 hingga 0,5 gigaton karbon per tahun secara global.[191]
Peran dalam pendauran kimia
Arkea mendaur ulang unsur-unsur seperti karbon, nitrogen dan belerang di berbagai habitat mereka.[192] Arkea melaksanakan banyak langkah dalam siklus nitrogen, yang meliputi reaksi yang dapat menghapus nitrogen dari ekosistem (seperti respirasi berbasis nitrat dan denitrifikasi) serta proses yang memperkenalkan nitrogen (seperti asimilasi nitrat dan fiksasi nitrogen).[193][194] Para peneliti baru-baru ini menemukan keterlibatan arkea pada reaksi oksidasi amonia. Reaksi-reaksi ini sangat penting dalam lautan.[136][195] Arkea juga tampaknya penting untuk oksidasi amonia di tanah. Mereka menghasilkan nitrit, dan mikrob lainnya mengoksidasi ke nitrat. Tumbuhan dan organisme lain mengkonsumsi nitrat.[196]
Pada daur belerang, arkea yang tumbuh dengan mengoksidasi senyawa sulfur melepaskan elemen ini dari batuan, membuatnya tersedia untuk organisme lain. Namun, arkea yang melakukan hal ini, seperti Sulfolobus, menghasilkan asam sulfat sebagai produk limbah, dan pertumbuhan organisme ini di tambang yang ditinggalkan dapat berkontribusi pada air asam tambang dan kerusakan lingkungan lainnya.[197]
Dalam siklus karbon, arkea metanogen menghapus hidrogen dan memainkan peran penting dalam pembusukan bahan organik dengan populasi mikroorganisme yang berperan sebagai dekomposer dalam ekosistem anaerob, seperti sedimen, rawa-rawa, dan pengolahan air limbah.[198]
Interaksi dengan organisme lain
Interaksi yang dikarakterisasi dengan baik antara arkea dan organisme lainnya bersifat mutual atau komensal. Tidak ada contoh yang jelas dan dikenal dari arkea yang bersifat patogenik atau parasitik.[199][200] Namun, beberapa spesies metanogen diperkirakan terlibat dalam infeksi di mulut,[201][202] dan Nanoarchaeum equitans mungkin merupakan parasit bagi spesies arkea lain karena hanya bertahan dan bereproduksi dalam sel-sel Crenarchaeon Ignicoccus hospitalis,[140] dan tampaknya tidak memberikan manfaat bagi inangnya.[203] Metanogen berperan sebagai mitra utama yang mendukung proses fermentasi bakteri yang didasarkan pada sintrofi yang bekerja dalam kondisi kekurangan energi.[204]
Mutualisme
Salah satu contoh mutualisme yang dipahami dengan baik adalah interaksi antara protozoa dan arkea metanogen dalam saluran pencernaan hewan yang mencerna selulosa, seperti ruminansia dan rayap.[205] Dalam lingkungan anaerobik ini, protozoa memecah selulosa tanaman untuk memperoleh energi. Proses ini melepaskan hidrogen sebagai produk limbah, tetapi hidrogen dalam tingkat tinggi mengurangi produksi energi. Ketika metanogen mengubah hidrogen menjadi metana, protozoa diuntungkan dengan lebih banyak energi.[206]
Dalam protozoa anaerobik, seperti Plagiopyla frontata, arkea berada di dalam protozoa dan mengonsumsi hidrogen yang diproduksi di hidrogenosom mereka.[207][208] Arkea juga berasosiasi dengan organisme yang lebih besar. Sebagai contoh, arkea laut Cenarchaeum symbiosum hidup dalam (merupakan endosimbion dari) spons Axinella mexicana.[209]
Komensalisme
Arkea juga bisa menjadi komensal, yang diuntungkan dari asosiasi tanpa membantu atau merugikan organisme lainnya. Sebagai contoh, metanogen Methanobrevibacter smithii adalah arkea paling umum dalam flora manusia, yang menyusun sekitar satu dari sepuluh dari semua prokariota dalam usus manusia.[210] Pada rayap dan manusia, metanogen ini mungkin sebenarnya mutualis, berinteraksi dengan mikrob lain dalam usus untuk membantu pencernaan.[211] Dalam tubuh manusia, arkea ditemukan di usus, rongga mulut, dan kulit.[212] Komunitas arkea juga berhubungan dengan berbagai organisme lain, seperti di permukaan karang,[213] dan di wilayah tanah yang mengelilingi akar tanaman (rizosfer).[214][215]
Peran dalam teknologi dan industri
Arkea ekstremofili, terutama yang tahan terhadap panas atau keasaman dan alkalinitas ekstrem, merupakan sumber enzim yang berfungsi di bawah kondisi yang keras.[216][217] Enzim-enzim ini memiliki banyak kegunaan. Misalnya, DNA polimerase termostabil, seperti Pfu DNA polimerase dari Pyrococcus furiosus, merevolusi biologi molekuler dengan memungkinkan reaksi berantai polimerase digunakan dalam penelitian sebagai teknik sederhana dan cepat untuk kloning DNA. Dalam industri, amilase, galaktosidase, dan pullulanase dari spesies Pyrococcus yang lain yang berfungsi pada suhu di atas 100 °C (212 °F) memungkinkan pengolahan makanan pada suhu tinggi, seperti produksi susu rendah laktosa dan whey.[218] Enzim dari arkea termofilik ini juga cenderung sangat stabil dalam pelarut organik, memungkinkan penggunaannya dalam proses ramah lingkungan dalam kimia hijau yang menyintesis senyawa organik.[217] Stabilitas ini membuatnya lebih mudah untuk digunakan dalam biologi struktural. Akibatnya, enzim dari arkea ekstremofil yang ekuivalen dengan enzim bakteri atau eukariotik sering digunakan dalam studi struktural.[219]
Berbeda dengan berbagai aplikasi enzim arkea, penggunaan organisme-organisme arkea sendiri di bidang bioteknologi kurang dikembangkan. Arkea metanogen adalah bagian penting dari pengolahan limbah karena mereka adalah bagian dari komunitas mikroorganisme yang melaksanakan pencernaan anaerobik dan menghasilkan biogas.[220] Dalam pengolahan mineral, arkea asidofilik dipandang menjanjikan untuk ekstraksi logam dari bijih, termasuk emas, kobalt dan tembaga.[221]
Arkea menghasilkan antibiotik kelas baru yang berpotensi berguna. Beberapa arkeosin ini telah dikarakterisasi, tetapi ratusan lainnya diyakini ada, terutama dalam Haloarchaea dan Sulfolobus. Senyawa-senyawa ini memiliki struktur yang berbeda dari antibiotik bakteri sehingga mereka mungkin memiliki cara kerja yang baru. Selain itu, mereka dapat memungkinkan pembuatan penanda yang dapat diseleksi yang baru untuk digunakan dalam biologi molekuler arkea.[222]
Lihat pula
Referensi
- ^ a b Woese CR, Kandler O, Wheelis ML (1990). "Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya". Proceedings of the National Academy of Sciences of the United States of America. 87 (12): 4576–9. Bibcode:1990PNAS...87.4576W. doi:10.1073/pnas.87.12.4576. PMC 54159 . PMID 2112744.
- ^ a b "Taxa above the rank of class". List of Prokaryotic names with Standing in Nomenclature. Diakses tanggal 8 August 2017.
- ^ Cavalier-Smith T (2014). "The neomuran revolution and phagotrophic origin of eukaryotes and cilia in the light of intracellular coevolution and a revised tree of life". Cold Spring Harb. Perspect. Biol. 6 (9): a016006. doi:10.1101/cshperspect.a016006. PMC 4142966 . PMID 25183828.
- ^ Petitjean, C., Deschamps, P., López-García, P., and Moreira, D. (2014). "Rooting the Domain archaea by phylogenomic analysis supports the foundation of the new kingdom proteoarchaeota". Genome Biol. Evol. 7 (1): 191–204. doi:10.1093/gbe/evu274. PMC 4316627 . PMID 25527841.
- ^ a b "NCBI taxonomy page on Archaea".
- ^ Staley JT (2006). "The bacterial species dilemma and the genomic-phylogenetic species concept". Philosophical Transactions of the Royal Society B. 361 (1475): 1899–909. doi:10.1098/rstb.2006.1914. PMC 1857736 . PMID 17062409.
- ^ Zuckerkandl E, Pauling L; Pauling (1965). "Molecules as documents of evolutionary history". J. Theor. Biol. 8 (2): 357–66. doi:10.1016/0022-5193(65)90083-4. PMID 5876245.
- ^ Parks DH, Chuvochina M, Waite DW, Rinke C, Skarshewski A, Chaumeil PA, Hugenholtz P (November 2018). "A standardized bacterial taxonomy based on genome phylogeny substantially revises the tree of life". Nature Biotechnology. 36 (10): 996–1004. doi:10.1038/nbt.4229. PMID 30148503.
- ^ a b c d Woese, C. R.; Fox, G. E. (1 November 1977). "Phylogenetic structure of the prokaryotic domain: The primary kingdoms". Proceedings of the National Academy of Sciences. 74 (11): 5088–5090. doi:10.1073/pnas.74.11.5088. ISSN 0027-8424. PMC 432104 . PMID 270744.
- ^ Pace NR (Mei 2006). "Time for a change". Nature. 441 (7091): 289. Bibcode:2006Natur.441..289P. doi:10.1038/441289a. PMID 16710401.
- ^ Sapp J (2009). The new foundations of evolution: on the tree of life. New York: Oxford University Press. ISBN 978-0-19-973438-2.
- ^ archaea. (2008). In Merriam-Webster Online Dictionary. Retrieved July 1, 2008, from http://www.merriam-webster.com/dictionary/archaea
- ^ Magrum LJ, Luehrsen KR, Woese CR; Luehrsen; Woese (1978). "Are extreme halophiles actually "bacteria"?". J Mol Evol. 11 (1): 1–10. doi:10.1007/bf01768019. PMID 660662.
- ^ Stetter KO. (1996). "Hyperthermophiles in the history of life". Ciba Found Symp. 202: 1–10. PMID 9243007.
- ^ a b c DeLong EF (1998). "Everything in moderation: archaea as 'non-extremophiles'". Current Opinion in Genetics & Development. 8 (6): 649–54. doi:10.1016/S0959-437X(98)80032-4. PMID 9914204.
- ^ Theron J, Cloete TE; Cloete (2000). "Molecular techniques for determining microbial diversity and community structure in natural environments". Crit. Rev. Microbiol. 26 (1): 37–57. doi:10.1080/10408410091154174. PMID 10782339.
- ^ Schmidt TM (2006). "The maturing of microbial ecology" (PDF). Int. Microbiol. 9 (3): 217–23. PMID 17061212.
- ^ Gevers D, Dawyndt P, Vandamme P; et al. (2006). "Stepping stones towards a new prokaryotic taxonomy". Philosophical Transactions of the Royal Society B. 361 (1475): 1911–6. doi:10.1098/rstb.2006.1915. PMC 1764938 . PMID 17062410.
- ^ a b Robertson CE, Harris JK, Spear JR, Pace NR; Harris; Spear; Pace (2005). "Phylogenetic diversity and ecology of environmental Archaea". Current Opinion in Microbiology. 8 (6): 638–42. doi:10.1016/j.mib.2005.10.003. PMID 16236543.
- ^ Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO.; Hohn; Rachel; Fuchs; Wimmer; Stetter (2002). "A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont". Nature. 417 (6884): 27–8. Bibcode:2002Natur.417...63H. doi:10.1038/417063a. PMID 11986665.
- ^ Barns SM, Delwiche CF, Palmer JD, Pace NR; Delwiche; Palmer; Pace (1996). "Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences". Proceedings of the National Academy of Sciences of the United States of America. 93 (17): 9188–93. Bibcode:1996PNAS...93.9188B. doi:10.1073/pnas.93.17.9188. PMC 38617 . PMID 8799176.
- ^ Elkins JG, Podar M, Graham DE; et al. (June 2008). "A korarchaeal genome reveals insights into the evolution of the Archaea". Proceedings of the National Academy of Sciences of the United States of America. 105 (23): 8102–7. Bibcode:2008PNAS..105.8102E. doi:10.1073/pnas.0801980105. PMC 2430366 . PMID 18535141.
- ^ Baker, B.J., Tyson, G.W., Webb, R.I., Flanagan, J., Hugenholtz, P. and Banfield, J.F. (2006). "Lineages of acidophilic Archaea revealed by community genomic analysis. Science". Science. 314 (6884): 1933–1935. Bibcode:2006Sci...314.1933B. doi:10.1126/science.1132690. PMID 17185602.
- ^ Baker BJ, Comolli LR, Dick GJ; et al. (May 2010). "Enigmatic, ultrasmall, uncultivated Archaea". Proceedings of the National Academy of Sciences of the United States of America. 107 (19): 8806–11. Bibcode:2010PNAS..107.8806B. doi:10.1073/pnas.0914470107. PMC 2889320 . PMID 20421484.
- ^ Guy, L; Ettema, TJ (19 December 2011). "The archaeal 'TACK' superphylum and the origin of eukaryotes". Trends Microbiol. 19 (12): 580–587. doi:10.1016/j.tim.2011.09.002. PMID 22018741.
- ^ a b Zaremba-Niedzwiedzka, K; et al. (2017). "Asgard archaea illuminate the origin of eukaryotic cellular complexity". Nature. 541: 353–358. doi:10.1038/nature21031.
- ^ Williams TA, Szöllősi GJ, Spang A, Foster PG, Heaps SE, Boussau B, et al. (June 2017). "Integrative modeling of gene and genome evolution roots the archaeal tree of life". Proceedings of the National Academy of Sciences of the United States of America. 114 (23): E4602–E4611. doi:10.1073/pnas.1618463114. PMC 5468678 . PMID 28533395.
- ^ Castelle CJ, Banfield JF (2018). "Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life". Cell. 172 (6): 1181–1197. doi:10.1016/j.cell.2018.02.016 . PMID 29522741.
- ^ Seitz KW, Dombrowski N, Eme L, Spang A, Lombard J, Sieber JR, et al. (April 2019). "Asgard archaea capable of anaerobic hydrocarbon cycling". Nature Communications. 10 (1): 1822. Bibcode:2019NatCo..10.1822S. doi:10.1038/s41467-019-09364-x. PMC 6478937 . PMID 31015394.
- ^ de Queiroz K (2005). "Ernst Mayr and the modern concept of species". Proceedings of the National Academy of Sciences of the United States of America. 102 (Suppl 1): 6600–7. Bibcode:2005PNAS..102.6600D. doi:10.1073/pnas.0502030102. PMC 1131873 . PMID 15851674.
- ^ Eppley JM, Tyson GW, Getz WM, Banfield JF; Tyson; Getz; Banfield (2007). "Genetic exchange across a species boundary in the archaeal genus ferroplasma". Genetics. 177 (1): 407–16. doi:10.1534/genetics.107.072892. PMC 2013692 . PMID 17603112.
- ^ Papke RT, Zhaxybayeva O, Feil EJ, Sommerfeld K, Muise D, Doolittle WF; Zhaxybayeva; Feil; Sommerfeld; Muise; Doolittle (2007). "Searching for species in haloarchaea". Proceedings of the National Academy of Sciences of the United States of America. 104 (35): 14092–7. Bibcode:2007PNAS..10414092P. doi:10.1073/pnas.0706358104. PMC 1955782 . PMID 17715057.
- ^ Kunin V, Goldovsky L, Darzentas N, Ouzounis CA; Goldovsky; Darzentas; Ouzounis (2005). "The net of life: reconstructing the microbial phylogenetic network". Genome Res. 15 (7): 954–9. doi:10.1101/gr.3666505. PMC 1172039 . PMID 15965028.
- ^ Hugenholtz P (2002). "Exploring prokaryotic diversity in the genomic era". Genome Biol. 3 (2): REVIEWS0003. doi:10.1186/gb-2002-3-2-reviews0003. PMC 139013 . PMID 11864374.
- ^ Rappé MS, Giovannoni SJ; Giovannoni (2003). "The uncultured microbial majority". Annu. Rev. Microbiol. 57: 369–94. doi:10.1146/annurev.micro.57.030502.090759. PMID 14527284.
- ^ "Age of the Earth". U.S. Geological Survey. 1997. Diarsipkan dari versi asli tanggal 23 December 2005. Diakses tanggal 2006-01-10.
- ^ Dalrymple, G. Brent (2001). "The age of the Earth in the twentieth century: a problem (mostly) solved". Special Publications, Geological Society of London. 190 (1): 205–221. Bibcode:2001GSLSP.190..205D. doi:10.1144/GSL.SP.2001.190.01.14.
- ^ Manhesa, Gérard; Allègre, Claude J.; Dupréa, Bernard & Hamelin, Bruno (1980). "Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics". Earth and Planetary Science Letters. 47 (3): 370–382. Bibcode:1980E&PSL..47..370M. doi:10.1016/0012-821X(80)90024-2.
- ^ de Duve, Christian (October 1995). "The Beginnings of Life on Earth". American Scientist. Diakses tanggal 15 January 2014.
- ^ Timmer, John (4 September 2012). "3.5 billion year old organic deposits show signs of life". Ars Technica. Diakses tanggal 15 January 2014.
- ^ Yoko Ohtomo, Takeshi Kakegawa, Akizumi Ishida, Toshiro Nagase, Minik T. Rosing (8 December 2013). "Evidence for biogenic graphite in early Archaean Isua metasedimentary rocks". Nature Geoscience. doi:10.1038/ngeo2025. Diakses tanggal 9 Dec 2013.
- ^ Borenstein, Seth (13 November 2013). "Oldest fossil found: Meet your microbial mom". AP News. Diakses tanggal 15 November 2013.
- ^ Noffke, Nora; Christian, Daniel; Wacey, David; Hazen, Robert M. (8 November 2013). "Microbially Induced Sedimentary Structures Recording an Ancient Ecosystem in the ca. 3.48 Billion-Year-Old Dresser Formation, Pilbara, Western Australia". Astrobiology (journal). 13 (12): 1103–24. Bibcode:2013AsBio..13.1103N. doi:10.1089/ast.2013.1030. PMC 3870916 . PMID 24205812. Diakses tanggal 15 November 2013.
- ^ Borenstein, Seth (19 October 2015). "Hints of life on what was thought to be desolate early Earth". Excite. Yonkers, NY: Mindspark Interactive Network. Associated Press. Diakses tanggal 2015-10-20.
- ^ Bell, Elizabeth A.; Boehnike, Patrick; Harrison, T. Mark; et al. (19 October 2015). "Potentially biogenic carbon preserved in a 4.1 billion-year-old zircon" (PDF). Proc. Natl. Acad. Sci. U.S.A. Washington, D.C.: National Academy of Sciences. 112 (47): 14518–21. doi:10.1073/pnas.1517557112. ISSN 1091-6490. PMC 4664351 . PMID 26483481. Diakses tanggal 2015-10-20. Early edition, published online before print.
- ^ Schopf J (2006). "Fossil evidence of Archaean life" (PDF). Philosophical Transactions of the Royal Society B. 361 (1470): 869–85. doi:10.1098/rstb.2006.1834. PMC 1578735 . PMID 16754604.[pranala nonaktif permanen]
- ^ Chappe B, Albrecht P, Michaelis W; Albrecht; Michaelis (July 1982). "Polar Lipids of Archaebacteria in Sediments and Petroleums". Science. 217 (4554): 65–66. Bibcode:1982Sci...217...65C. doi:10.1126/science.217.4554.65. PMID 17739984.
- ^ Brocks JJ, Logan GA, Buick R, Summons RE; Logan; Buick; Summons (1999). "Archean molecular fossils and the early rise of eukaryotes". Science. 285 (5430): 1033–6. doi:10.1126/science.285.5430.1033. PMID 10446042.
- ^ Rasmussen B, Fletcher IR, Brocks JJ, Kilburn MR; Fletcher; Brocks; Kilburn (October 2008). "Reassessing the first appearance of eukaryotes and cyanobacteria". Nature. 455 (7216): 1101–4. Bibcode:2008Natur.455.1101R. doi:10.1038/nature07381. PMID 18948954.
- ^ Hahn, Jürgen; Pat Haug (1986). "Traces of Archaebacteria in ancient sediments". System Applied Microbiology. 7 (Archaebacteria '85 Proceedings): 178–83. doi:10.1016/S0723-2020(86)80002-9.
- ^ Wang M, Yafremava LS, Caetano-Anollés D, Mittenthal JE, Caetano-Anollés G; Yafremava; Caetano-Anollés; Mittenthal; Caetano-Anollés (2007). "Reductive evolution of architectural repertoires in proteomes and the birth of the tripartite world". Genome Res. 17 (11): 1572–85. doi:10.1101/gr.6454307. PMC 2045140 . PMID 17908824.
- ^ Woese CR, Gupta R; Gupta (1981). "Are archaebacteria merely derived 'prokaryotes'?". Nature. 289 (5793): 95–6. Bibcode:1981Natur.289...95W. doi:10.1038/289095a0. PMID 6161309.
- ^ a b c >Woese C (1998). "The universal ancestor". Proceedings of the National Academy of Sciences of the United States of America. 95 (12): 6854–9. Bibcode:1998PNAS...95.6854W. doi:10.1073/pnas.95.12.6854. PMC 22660 . PMID 9618502.
- ^ a b Kandler O. The early diversification of life and the origin of the three domains: A proposal. In: Wiegel J, Adams WW, editors. Thermophiles: The keys to molecular evolution and the origin of life? Athens: Taylor and Francis, 1998: 19-31.
- ^ Gribaldo S, Brochier-Armanet C; Brochier-Armanet (2006). "The origin and evolution of Archaea: a state of the art". Philosophical Transactions of the Royal Society B. 361 (1470): 1007–22. doi:10.1098/rstb.2006.1841. PMC 1578729 . PMID 16754611. Diarsipkan dari versi asli tanggal 2012-06-04. Diakses tanggal 2015-08-15.
- ^ a b Woese CR (1 March 1994). "There must be a prokaryote somewhere: microbiology's search for itself". Microbiol. Rev. 58 (1): 1–9. PMC 372949 . PMID 8177167.
- ^ Information is from Willey JM, Sherwood LM, Woolverton CJ. Microbiology 7th ed. (2008), Ch. 19 pp. 474–475, except where noted.
- ^ Heimerl, Thomas; Flechsler, Jennifer; Pickl, Carolin; Heinz, Veronika; Salecker, Benjamin; Zweck, Josef; Wanner, Gerhard; Geimer, Stefan; Samson, Rachel Y. (2017-06-13). "A Complex Endomembrane System in the Archaeon Ignicoccus hospitalis Tapped by Nanoarchaeum equitans". Frontiers in Microbiology. 8: 1072. doi:10.3389/fmicb.2017.01072. ISSN 1664-302X. PMC 5468417 . PMID 28659892.
- ^ Jurtshuk, Peter (1996). Medical Microbiology (edisi ke-4). Galveston (TX): University of Texas Medical Branch at Galveston. Diakses tanggal 5 November 2014.
- ^ Howland, John L. (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. hlm. 25–30. ISBN 0-19-511183-4.
- ^ a b c Cavicchioli, Ricardo (January 2011). "Archaea- timeline of the third domain". Nature Reviews Microbiology. 9 (1): 51–61. doi:10.1038/nrmicro2482. PMID 21132019. Diakses tanggal 5 November 2014.
- ^ Gupta, Radhey S.; Shami, Ali (2011-02). "Molecular signatures for the Crenarchaeota and the Thaumarchaeota". Antonie van Leeuwenhoek (dalam bahasa Inggris). 99 (2): 133–157. doi:10.1007/s10482-010-9488-3. ISSN 0003-6072.
- ^ Gao, Beile; Gupta, Radhey S (2007). "Phylogenomic analysis of proteins that are distinctive of Archaea and its main subgroups and the origin of methanogenesis". BMC Genomics. 8 (1): 86. doi:10.1186/1471-2164-8-86. PMC 1852104 . PMID 17394648.
- ^ Gupta, Radhey S.; Naushad, Sohail; Baker, Sheridan (2015-03-01). "Phylogenomic analyses and molecular signatures for the class Halobacteria and its two major clades: a proposal for division of the class Halobacteria into an emended order Halobacteriales and two new orders, Haloferacales ord. nov. and Natrialbales ord. nov., containing the novel families Haloferacaceae fam. nov. and Natrialbaceae fam. nov". International Journal of Systematic and Evolutionary Microbiology (dalam bahasa Inggris). 65 (Pt_3): 1050–1069. doi:10.1099/ijs.0.070136-0. ISSN 1466-5026.
- ^ Deppenmeier, U. (2002). "The unique biochemistry of methanogenesis". PubMed. 71: 223–283. doi:10.1016/s0079-6603(02)71045-3. PMID 12102556. Diakses tanggal 5 November 2014.
- ^ Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P; Doerks; von Mering; Creevey; Snel; Bork (2006). "Toward automatic reconstruction of a highly resolved tree of life". Science. 311 (5765): 1283–7. Bibcode:2006Sci...311.1283C. doi:10.1126/science.1123061. PMID 16513982.
- ^ Koonin EV, Mushegian AR, Galperin MY, Walker DR; Mushegian; Galperin; Walker (1997). "Comparison of archaeal and bacterial genomes: computer analysis of protein sequences predicts novel functions and suggests a chimeric origin for the archaea". Mol Microbiol. 25 (4): 619–637. doi:10.1046/j.1365-2958.1997.4821861.x. PMID 9379893.
- ^ a b c d e Gupta R. S. (1998). "Protein phylogenies and signature sequences: A reappraisal of evolutionary relationships among archaebacteria, eubacteria, and eukaryotes". Microbiol. Mol. Biol. Rev. 62 (4): 1435–1491. PMC 98952 . PMID 9841678.
- ^ Koch AL (2003). "Were Gram-positive rods the first bacteria?". Trends Microbiol. 11 (4): 166–170. doi:10.1016/S0966-842X(03)00063-5. PMID 12706994.
- ^ a b c Gupta R.S. (1998). "What are archaebacteria: life's third domain or monoderm prokaryotes related to gram-positive bacteria? A new proposal for the classification of prokaryotic organisms". Mol. Microbiol. 29 (3): 695–708. doi:10.1046/j.1365-2958.1998.00978.x. PMID 9723910.
- ^ Gogarten, Johann Peter (1994). "Which is the Most Conserved Group of Proteins? Homology - Orthology, Paralogy, Xenology and the Fusion of Independent Lineages". Journal of Molecular Evolution. 39 (5): 541–543. doi:10.1007/bf00173425. PMID 7807544.
- ^ Brown JR, Masuchi Y, Robb FT, Doolittle WF; Masuchi; Robb; Doolittle (1994). "Evolutionary relationships of bacterial and archaeal glutamine synthetase genes". J Mol Evol. 38 (6): 566–576. doi:10.1007/BF00175876. PMID 7916055.
- ^ Katz, Laura A. (2015-09-26). "Recent events dominate interdomain lateral gene transfers between prokaryotes and eukaryotes and, with the exception of endosymbiotic gene transfers, few ancient transfer events persist". Philosophical Transactions of the Royal Society B: Biological Sciences (dalam bahasa Inggris). 370 (1678): 20140324. doi:10.1098/rstb.2014.0324. ISSN 0962-8436. PMC 4571564 . PMID 26323756.
- ^ a b c Gupta R.S. (2000). "The natural evolutionary relationships among prokaryotes". Crit. Rev. Microbiol. 26 (2): 111–131. doi:10.1080/10408410091154219. PMID 10890353.
- ^ Gupta RS. Molecular Sequences and the Early History of Life. In: Sapp J, editor. Microbial Phylogeny and Evolution: Concepts and Controversies. New York: Oxford University Press, 2005: 160-183.
- ^ Cavalier-Smith T (2002). "The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification". Int J Syst Evol Microbiol. 52 (1): 7–76. PMID 11837318.
- ^ Valas RE, Bourne PE; Bourne (2011). "The origin of a derived superkingdom: how a Gram-positive bacterium crossed the desert to become an archaeon". Biol Direct. 6: 16. doi:10.1186/1745-6150-6-16. PMC 3056875 . PMID 21356104.
- ^ Skophammer RG, Herbold CW, Rivera MC, Servin JA, Lake JA; Herbold; Rivera; Servin; Lake (2006). "Evidence that the root of the tree of life is not within the Archaea". Mol Biol Evol. 23 (9): 1648–1651. doi:10.1093/molbev/msl046. PMID 16801395.
- ^ Eme, Laura; Spang, Anja; Lombard, Jonathan; Stairs, Courtney W.; Ettema, Thijs J. G. (2017-12). "Archaea and the origin of eukaryotes". Nature Reviews Microbiology (dalam bahasa Inggris). 15 (12): 711–723. doi:10.1038/nrmicro.2017.133. ISSN 1740-1526.
- ^ a b Lake JA (January 1988). "Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences". Nature. 331 (6152): 184–6. Bibcode:1988Natur.331..184L. doi:10.1038/331184a0. PMID 3340165.
- ^ Nelson KE, Clayton RA, Gill SR; et al. (1999). "Evidence for lateral gene transfer between Archaea and bacteria from genome sequence of Thermotoga maritima". Nature. 399 (6734): 323–9. Bibcode:1999Natur.399..323N. doi:10.1038/20601. PMID 10360571.
- ^ Gouy M, Li WH; Li (May 1989). "Phylogenetic analysis based on rRNA sequences supports the archaebacterial rather than the eocyte tree". Nature. 339 (6220): 145–7. Bibcode:1989Natur.339..145G. doi:10.1038/339145a0. PMID 2497353.
- ^ Yutin N, Makarova KS, Mekhedov SL, Wolf YI, Koonin EV; Makarova; Mekhedov; Wolf; Koonin (May 2008). "The deep archaeal roots of eukaryotes". Mol. Biol. Evol. 25 (8): 1619–30. doi:10.1093/molbev/msn108. PMC 2464739 . PMID 18463089.
- ^ Williams, Tom A.; Foster, Peter G.; Cox, Cymon J.; Embley, T. Martin (December 2013). "An archaeal origin of eukaryotes supports only two primary domains of life". Nature. 504 (7479): 231–236. Bibcode:2013Natur.504..231W. doi:10.1038/nature12779. PMID 24336283.
- ^ Zimmer, Carl (May 6, 2015). "Under the Sea, a Missing Link in the Evolution of Complex Cells". New York Times. Diakses tanggal May 6, 2015.
- ^ Spang, Anja; Saw, Jimmy H.; Jørgensen, Steffen L.; Zaremba-Niedzwiedzka, Katarzyna; Martijn, Joran; Lind, Anders E.; Eijk, Roel van; Schleper, Christa; Guy, Lionel (2015). "Complex archaea that bridge the gap between prokaryotes and eukaryotes". Nature. 521 (7551): 173–179. Bibcode:2015Natur.521..173S. doi:10.1038/nature14447. PMC 4444528 . PMID 25945739.
- ^ Seitz, Kiley W.; Lazar, Cassandre S.; Hinrichs, Kai-Uwe; Teske, Andreas P.; Baker, Brett J. (2016). "Genomic reconstruction of a novel, deeply branched sediment archaeal phylum with pathways for acetogenesis and sulfur reduction". ISME Journal. 10 (7): 1696–1705. doi:10.1038/ismej.2015.233. PMID 26824177.
- ^ MacLeod, Fraser; Kindler, Gareth S.; Wong, Hon Lun; Chen, Ray; Burns, Brendan P. (2019). "Asgard archaea: Diversity, function, and evolutionary implications in a range of microbiomes". AIMS Microbiology (dalam bahasa Inggris). 5 (1): 48. doi:10.3934/microbiol.2019.1.48. PMC 6646929 . PMID 31384702.
- ^ Zimmer, Carl (2020-01-15). "This Strange Microbe May Mark One of Life's Great Leaps". The New York Times (dalam bahasa Inggris). ISSN 0362-4331. Diakses tanggal 2020-11-04.
- ^ Imachi, Hiroyuki; Nobu, Masaru K.; Nakahara, Nozomi; Morono, Yuki; Ogawara, Miyuki; Takaki, Yoshihiro; Takano, Yoshinori; Uematsu, Katsuyuki; Ikuta, Tetsuro (2020-01-23). "Isolation of an archaeon at the prokaryote–eukaryote interface". Nature (dalam bahasa Inggris). 577 (7791): 519–525. doi:10.1038/s41586-019-1916-6. ISSN 0028-0836. PMC 7015854 . PMID 31942073.
- ^ a b c d Krieg, Noel (2005). Bergey's Manual of Systematic Bacteriology. US: Springer. hlm. 21–6. ISBN 978-0-387-24143-2.
- ^ Barns, Sue and Burggraf, Siegfried. (1997) Crenarchaeota. Version 1 January 1997. in The Tree of Life Web Project
- ^ Stoeckenius W (1 October 1981). "Walsby's square bacterium: fine structure of an orthogonal procaryote". J. Bacteriol. 148 (1): 352–60. PMC 216199 . PMID 7287626.
- ^ Walsby, A.E. (1980). "A square bacterium". Nature. 283 (5742): 69–71. Bibcode:1980Natur.283...69W. doi:10.1038/283069a0.
- ^ Hara F, Yamashiro K, Nemoto N; et al. (2007). "An actin homolog of the archaeon Thermoplasma acidophilum that retains the ancient characteristics of eukaryotic actin". J. Bacteriol. 189 (5): 2039–45. doi:10.1128/JB.01454-06. PMC 1855749 . PMID 17189356.
- ^ Trent JD, Kagawa HK, Yaoi T, Olle E, Zaluzec NJ; Kagawa; Yaoi; Olle; Zaluzec (1997). "Chaperonin filaments: the archaeal cytoskeleton?". Proceedings of the National Academy of Sciences of the United States of America. 94 (10): 5383–8. Bibcode:1997PNAS...94.5383T. doi:10.1073/pnas.94.10.5383. PMC 24687 . PMID 9144246.
- ^ Hixon WG, Searcy DG; Searcy (1993). "Cytoskeleton in the archaebacterium Thermoplasma acidophilum? Viscosity increase in soluble extracts". BioSystems. 29 (2–3): 151–60. doi:10.1016/0303-2647(93)90091-P. PMID 8374067.
- ^ a b Golyshina OV, Pivovarova TA, Karavaiko GI; et al. (1 May 2000). "Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea". Int. J. Syst. Evol. Microbiol. 50 (3): 997–1006. doi:10.1099/00207713-50-3-997. PMID 10843038.[pranala nonaktif permanen]
- ^ Hall-Stoodley L, Costerton JW, Stoodley P; Costerton; Stoodley (2004). "Bacterial biofilms: from the natural environment to infectious diseases". Nature Reviews Microbiology. 2 (2): 95–108. doi:10.1038/nrmicro821. PMID 15040259.
- ^ Kuwabara T, Minaba M, Iwayama Y; et al. (November 2005). "Thermococcus coalescens sp. nov., a cell-fusing hyperthermophilic archaeon from Suiyo Seamount". Int. J. Syst. Evol. Microbiol. 55 (Pt 6): 2507–14. doi:10.1099/ijs.0.63432-0. PMID 16280518.[pranala nonaktif permanen]
- ^ Nickell S, Hegerl R, Baumeister W, Rachel R; Hegerl; Baumeister; Rachel (2003). "Pyrodictium cannulae enter the periplasmic space but do not enter the cytoplasm, as revealed by cryo-electron tomography". J. Struct. Biol. 141 (1): 34–42. doi:10.1016/S1047-8477(02)00581-6. PMID 12576018.
- ^ Horn C, Paulmann B, Kerlen G, Junker N, Huber H; Paulmann; Kerlen; Junker; Huber (15 August 1999). "In vivo observation of cell division of anaerobic hyperthermophiles by using a high-intensity dark-field microscope". J. Bacteriol. 181 (16): 5114–8. PMC 94007 . PMID 10438790.
- ^ Rudolph C, Wanner G, Huber R; Wanner; Huber (May 2001). "Natural communities of novel archaea and bacteria growing in cold sulfurous springs with a string-of-pearls-like morphology". Appl. Environ. Microbiol. 67 (5): 2336–44. doi:10.1128/AEM.67.5.2336-2344.2001. PMC 92875 . PMID 11319120.
- ^ a b Thomas NA, Bardy SL, Jarrell KF; Bardy; Jarrell (2001). "The archaeal flagellum: a different kind of prokaryotic motility structure". FEMS Microbiol. Rev. 25 (2): 147–74. doi:10.1111/j.1574-6976.2001.tb00575.x. PMID 11250034.
- ^ Rachel R, Wyschkony I, Riehl S, Huber H; Wyschkony; Riehl; Huber (March 2002). "The ultrastructure of Ignicoccus: evidence for a novel outer membrane and for intracellular vesicle budding in an archaeon" (PDF). Archaea. 1 (1): 9–18. doi:10.1155/2002/307480. PMC 2685547 . PMID 15803654. Diarsipkan dari versi asli (PDF) tanggal 2009-02-24. Diakses tanggal 2015-08-24.
- ^ Sára M, Sleytr UB; Sleytr (2000). "S-Layer proteins". J. Bacteriol. 182 (4): 859–68. doi:10.1128/JB.182.4.859-868.2000. PMC 94357 . PMID 10648507.
- ^ Engelhardt H, Peters J; Peters (1998). "Structural research on surface layers: a focus on stability, surface layer homology domains, and surface layer-cell wall interactions". J Struct Biol. 124 (2–3): 276–302. doi:10.1006/jsbi.1998.4070. PMID 10049812.
- ^ a b Kandler, O; König, H (1998). "Cell wall polymers in Archaea (Archaebacteria)" (PDF). Cellular and Molecular Life Sciences (CMLS). 54 (4): 305–308. doi:10.1007/s000180050156.[pranala nonaktif permanen]
- ^ Howland, John L. (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. hlm. 32. ISBN 0-19-511183-4.
- ^ Gophna U, Ron EZ, Graur D; Ron; Graur (July 2003). "Bacterial type III secretion systems are ancient and evolved by multiple horizontal-transfer events". Gene. 312: 151–63. doi:10.1016/S0378-1119(03)00612-7. PMID 12909351.
- ^ Nguyen L, Paulsen IT, Tchieu J, Hueck CJ, Saier MH; Paulsen; Tchieu; Hueck; Saier Jr (April 2000). "Phylogenetic analyses of the constituents of Type III protein secretion systems". J. Mol. Microbiol. Biotechnol. 2 (2): 125–44. PMID 10939240.
- ^ Ng SY, Chaban B, Jarrell KF; Chaban; Jarrell (2006). "Archaeal flagella, bacterial flagella and type IV pili: a comparison of genes and posttranslational modifications". J. Mol. Microbiol. Biotechnol. 11 (3–5): 167–91. doi:10.1159/000094053. PMID 16983194.
- ^ Bardy SL, Ng SY, Jarrell KF; Ng; Jarrell (February 2003). "Prokaryotic motility structures". Microbiology (Reading, Engl.). 149 (Pt 2): 295–304. doi:10.1099/mic.0.25948-0. PMID 12624192.
- ^ a b Koga Y, Morii H; Morii (2007). "Biosynthesis of ether-type polar lipids in archaea and evolutionary considerations". Microbiol. Mol. Biol. Rev. 71 (1): 97–120. doi:10.1128/MMBR.00033-06. PMC 1847378 . PMID 17347520.
- ^ Youssefian, Sina; Rahbar, Nima; Van Dessel, Steven (2018-05-07). "Thermal conductivity and rectification in asymmetric archaeal lipid membranes". The Journal of Chemical Physics (dalam bahasa Inggris). 148 (17): 174901. doi:10.1063/1.5018589. ISSN 0021-9606.
- ^ "Archaea Basic Biology". Basic Biology. March 2018. Diakses tanggal 3 November 2020.
- ^ De Rosa M, Gambacorta A, Gliozzi A; Gambacorta; Gliozzi (1 March 1986). "Structure, biosynthesis, and physicochemical properties of archaebacterial lipids". Microbiol. Rev. 50 (1): 70–80. PMC 373054 . PMID 3083222.
- ^ Albers SV, van de Vossenberg JL, Driessen AJ, Konings WN; Van De Vossenberg; Driessen; Konings (September 2000). "Adaptations of the archaeal cell membrane to heat stress". Front. Biosci. 5: D813–20. doi:10.2741/albers. PMID 10966867.
- ^ Damsté JS, Schouten S, Hopmans EC, van Duin AC, Geenevasen JA; Schouten; Hopmans; Van Duin; Geenevasen (October 2002). "Crenarchaeol: the characteristic core glycerol dibiphytanyl glycerol tetraether membrane lipid of cosmopolitan pelagic crenarchaeota". J. Lipid Res. 43 (10): 1641–51. doi:10.1194/jlr.M200148-JLR200. PMID 12364548.
- ^ Koga Y, Morii H; Morii (November 2005). "Recent advances in structural research on ether lipids from arkea including comparative and physiological aspects". Biosci. Biotechnol. Biochem. 69 (11): 2019–34. doi:10.1271/bbb.69.2019. PMID 16306681.
- ^ Hanford MJ, Peeples TL; Peeples (January 2002). "Archaeal tetraether lipids: unique structures and applications". Appl. Biochem. Biotechnol. 97 (1): 45–62. doi:10.1385/ABAB:97:1:45. PMID 11900115.
- ^ Macalady JL, Vestling MM, Baumler D, Boekelheide N, Kaspar CW, Banfield JF; Vestling; Baumler; Boekelheide; Kaspar; Banfield (October 2004). "Tetraether-linked membrane monolayers in Ferroplasma spp: a key to survival in acid". Extremophiles. 8 (5): 411–9. doi:10.1007/s00792-004-0404-5. PMID 15258835.
- ^ a b c Valentine DL (2007). "Adaptations to energy stress dictate the ecology and evolution of the Archaea". Nature Reviews Microbiology. 5 (4): 316–23. doi:10.1038/nrmicro1619. PMID 17334387.
- ^ a b c Schäfer G, Engelhard M, Müller V; Engelhard; Müller (1 September 1999). "Bioenergetics of the Archaea". Microbiol. Mol. Biol. Rev. 63 (3): 570–620. PMC 103747 . PMID 10477309.
- ^ Zillig W (December 1991). "Comparative biochemistry of Archaea and Bacteria". Current Opinion in Genetics & Development. 1 (4): 544–51. doi:10.1016/S0959-437X(05)80206-0. PMID 1822288.
- ^ Romano A, Conway T; Conway (1996). "Evolution of carbohydrate metabolic pathways". Res Microbiol. 147 (6–7): 448–55. doi:10.1016/0923-2508(96)83998-2. PMID 9084754.
- ^ Koch A (1998). "How did bacteria come to be?". Adv Microb Physiol. Advances in Microbial Physiology. 40: 353–99. doi:10.1016/S0065-2911(08)60135-6. ISBN 9780120277407. PMID 9889982.
- ^ DiMarco AA, Bobik TA, Wolfe RS; Bobik; Wolfe (1990). "Unusual coenzymes of methanogenesis". Annu. Rev. Biochem. 59: 355–94. doi:10.1146/annurev.bi.59.070190.002035. PMID 2115763.
- ^ Klocke M, Nettmann E, Bergmann I; et al. (May 2008). "Characterization of the methanogenic Archaea within two-phase biogas reactor systems operated with plant biomass". Syst. Appl. Microbiol. 31 (3): 190–205. doi:10.1016/j.syapm.2008.02.003. PMID 18501543.
- ^ Based on PDB 1FBB. Data published in Subramaniam S, Henderson R; Henderson (August 2000). "Molecular mechanism of vectorial proton translocation by bacteriorhodopsin". Nature. 406 (6796): 653–7. doi:10.1038/35020614. PMID 10949309.
- ^ Mueller-Cajar O, Badger MR; Badger (August 2007). "New roads lead to Rubisco in archaebacteria". BioEssays. 29 (8): 722–4. doi:10.1002/bies.20616. PMID 17621634.
- ^ Berg IA, Kockelkorn D, Buckel W, Fuchs G; Kockelkorn; Buckel; Fuchs (December 2007). "A 3-hydroxypropionate/4-hydroxybutyrate autotrophic carbon dioxide assimilation pathway in Archaea". Science. 318 (5857): 1782–6. Bibcode:2007Sci...318.1782B. doi:10.1126/science.1149976. PMID 18079405.
- ^ Thauer RK (December 2007). "Microbiology. A fifth pathway of carbon fixation". Science. 318 (5857): 1732–3. doi:10.1126/science.1152209. PMID 18079388.
- ^ Bryant DA, Frigaard NU; Frigaard (November 2006). "Prokaryotic photosynthesis and phototrophy illuminated". Trends Microbiol. 14 (11): 488–96. doi:10.1016/j.tim.2006.09.001. PMID 16997562.
- ^ a b Könneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA; Bernhard; de la Torre; Walker; Waterbury; Stahl (September 2005). "Isolation of an autotrophic ammonia-oxidizing marine archaeon". Nature. 437 (7058): 543–6. Bibcode:2005Natur.437..543K. doi:10.1038/nature03911. PMID 16177789.
- ^ a b Francis CA, Beman JM, Kuypers MM; Beman; Kuypers (May 2007). "New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation". ISME J. 1 (1): 19–27. doi:10.1038/ismej.2007.8. PMID 18043610.
- ^ Lanyi JK (2004). "Bacteriorhodopsin". Annu. Rev. Physiol. 66: 665–88. doi:10.1146/annurev.physiol.66.032102.150049. PMID 14977418.
- ^ a b Allers T, Mevarech M; Mevarech (2005). "Archaeal genetics — the third way". Nature Reviews Genetics. 6 (1): 58–73. doi:10.1038/nrg1504. PMID 15630422.
- ^ Galagan JE, Nusbaum C, Roy A; et al. (April 2002). "The genome of M. acetivorans reveals extensive metabolic and physiological diversity". Genome Res. 12 (4): 532–42. doi:10.1101/gr.223902. PMC 187521 . PMID 11932238.
- ^ a b Waters E; et al. (2003). "The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism". Proceedings of the National Academy of Sciences of the United States of America. 100 (22): 12984–8. Bibcode:2003PNAS..10012984W. doi:10.1073/pnas.1735403100. PMC 240731 . PMID 14566062.
- ^ Schleper C, Holz I, Janekovic D, Murphy J, Zillig W; Holz; Janekovic; Murphy; Zillig (1 August 1995). "A multicopy plasmid of the extremely thermophilic archaeon Sulfolobus effects its transfer to recipients by mating". J. Bacteriol. 177 (15): 4417–26. PMC 177192 . PMID 7635827.
- ^ Sota M; Top EM (2008). "Horizontal Gene Transfer Mediated by Plasmids". Plasmids: Current Research and Future Trends. Caister Academic Press. isbn=978-1-904455-35-6.link)
- ^ Xiang X, Chen L, Huang X, Luo Y, She Q, Huang L; Chen; Huang; Luo; She; Huang (2005). "Sulfolobus tengchongensis spindle-shaped virus STSV1: virus-host interactions and genomic features". J. Virol. 79 (14): 8677–86. doi:10.1128/JVI.79.14.8677-8686.2005. PMC 1168784 . PMID 15994761.
- ^ Graham DE, Overbeek R, Olsen GJ, Woese CR; Overbeek; Olsen; Woese (2000). "An archaeal genomic signature". Proceedings of the National Academy of Sciences of the United States of America. 97 (7): 3304–8. Bibcode:2000PNAS...97.3304G. doi:10.1073/pnas.050564797. PMC 16234 . PMID 10716711.
- ^ a b Gaasterland T (1999). "Archaeal genomics". Current Opinion in Microbiology. 2 (5): 542–7. doi:10.1016/S1369-5274(99)00014-4. PMID 10508726.
- ^ Dennis PP (1997). "Ancient Ciphers: Translation in Archaea". Cell. 89 (7): 1007–10. doi:10.1016/S0092-8674(00)80288-3. PMID 9215623.
- ^ Werner F (September 2007). "Structure and function of archaeal RNA polymerases". Mol. Microbiol. 65 (6): 1395–404. doi:10.1111/j.1365-2958.2007.05876.x. PMID 17697097.
- ^ Aravind L, Koonin EV; Koonin (1999). "DNA-binding proteins and evolution of transcription regulation in the archaea". Nucleic Acids Res. 27 (23): 4658–70. doi:10.1093/nar/27.23.4658. PMC 148756 . PMID 10556324.
- ^ Lykke-Andersen J, Aagaard C, Semionenkov M, Garrett RA; Aagaard; Semionenkov; Garrett (September 1997). "Archaeal introns: splicing, intercellular mobility and evolution". Trends Biochem. Sci. 22 (9): 326–31. doi:10.1016/S0968-0004(97)01113-4. PMID 9301331.
- ^ Watanabe Y, Yokobori S, Inaba T; et al. (January 2002). "Introns in protein-coding genes in Archaea". FEBS Lett. 510 (1–2): 27–30. doi:10.1016/S0014-5793(01)03219-7. PMID 11755525.
- ^ Yoshinari S, Itoh T, Hallam SJ; et al. (August 2006). "Archaeal pre-mRNA splicing: a connection to hetero-oligomeric splicing endonuclease". Biochem. Biophys. Res. Commun. 346 (3): 1024–32. doi:10.1016/j.bbrc.2006.06.011. PMID 16781672.
- ^ Rosenshine, I; Tchelet, R; Mevarech, M. (1989). "The mechanism of DNA transfer in the mating system of an archaebacterium". Science. 245 (4924): 1387–1389. Bibcode:1989Sci...245.1387R. doi:10.1126/science.2818746. PMID 2818746.
- ^ a b c Fröls, S; Ajon, M; Wagner, M; Teichmann, D; Zolghadr, B; Folea, M; Boekema, EJ; Driessen, AJ; Schleper, C; et al. (2008). "UV-inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus is mediated by pili formation". Mol Microbiol. 70 (4): 938–52. doi:10.1111/j.1365-2958.2008.06459.x. PMID 18990182.
- ^ a b c Ajon, M; Fröls, S; van Wolferen, M; Stoecker, K; Teichmann, D; Driessen, AJ; Grogan, DW; Albers, SV; Schleper, C.; et al. (2011). "UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili". Mol Microbiol. 82 (4): 807–17. doi:10.1111/j.1365-2958.2011.07861.x. PMID 21999488.
- ^ Fröls, S; White, MF; Schleper, C. (2009). "Reactions to UV damage in the model archaeon Sulfolobus solfataricus". Biochem Soc Trans. 37 (1): 36–41. doi:10.1042/BST0370036. PMID 19143598.
- ^ Bernstein, H; Bernstein, C (2013). Bernstein, Carol, ed. Evolutionary Origin and Adaptive Function of Meiosis, Meiosis. InTech. ISBN 978-953-51-1197-9.
- ^ a b Blohs, Marcus; Mahnert, Alexander; Spang, Anja; Dombrowski, Nina; Krupovic, Mart; Klingl, Andreas (2019). Reference Module in Life Sciences (dalam bahasa Inggris). Elsevier. hlm. B9780128096338208844. doi:10.1016/b978-0-12-809633-8.20884-4. ISBN 978-0-12-809633-8.
- ^ a b c Krupovic, Mart; Cvirkaite-Krupovic, Virginija; Iranzo, Jaime; Prangishvili, David; Koonin, Eugene V. (2018-01). "Viruses of archaea: Structural, functional, environmental and evolutionary genomics". Virus Research (dalam bahasa Inggris). 244: 181–193. doi:10.1016/j.virusres.2017.11.025. PMC 5801132 . PMID 29175107.
- ^ Pietilä, Maija K.; Demina, Tatiana A.; Atanasova, Nina S.; Oksanen, Hanna M.; Bamford, Dennis H. (2014-06). "Archaeal viruses and bacteriophages: comparisons and contrasts". Trends in Microbiology (dalam bahasa Inggris). 22 (6): 334–344. doi:10.1016/j.tim.2014.02.007.
- ^ Principi, Nicola; Silvestri, Ettore; Esposito, Susanna (2019-05-08). "Advantages and Limitations of Bacteriophages for the Treatment of Bacterial Infections". Frontiers in Pharmacology. 10: 513. doi:10.3389/fphar.2019.00513. ISSN 1663-9812. PMC 6517696 . PMID 31139086.
- ^ Prangishvili, D. (2013). Brenner's Encyclopedia of Genetics (dalam bahasa Inggris). Elsevier. hlm. 295–298. doi:10.1016/b978-0-12-374984-0.01627-2. ISBN 978-0-08-096156-9.
- ^ Prangishvili D, Garrett RA; Garrett (2004). "Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses". Biochem. Soc. Trans. 32 (Pt 2): 204–8. doi:10.1042/BST0320204. PMID 15046572.
- ^ Pietilä MK, Roine E, Paulin L, Kalkkinen N, Bamford DH; Roine; Paulin; Kalkkinen; Bamford (March 2009). "An ssDNA virus infecting archaea; A new lineage of viruses with a membrane envelope". Mol. Microbiol. 72 (2): 307–19. doi:10.1111/j.1365-2958.2009.06642.x. PMID 19298373.
- ^ Mochizuki T, Krupovic M, Pehau-Arnaudet G, Sako Y, Forterre P, Prangishvili D; Krupovic; Pehau-Arnaudet; Sako; Forterre; Prangishvili (2012). "Archaeal virus with exceptional virion architecture and the largest single-stranded DNA genome". Proceedings of the National Academy of Sciences of the United States of America. 109 (33): 13386–13391. Bibcode:2012PNAS..10913386M. doi:10.1073/pnas.1203668109. PMC 3421227 . PMID 22826255.
- ^ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E; Díez-Villaseñor; García-Martínez; Soria (2005). "Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements". J. Mol. Evol. 60 (2): 174–82. doi:10.1007/s00239-004-0046-3. PMID 15791728.
- ^ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV; Grishin; Shabalina; Wolf; Koonin (2006). "A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action". Biol. Direct. 1: 7. doi:10.1186/1745-6150-1-7. PMC 1462988 . PMID 16545108.
- ^ a b Bernander R (1998). "Archaea and the cell cycle". Mol. Microbiol. 29 (4): 955–61. doi:10.1046/j.1365-2958.1998.00956.x. PMID 9767564.
- ^ Kelman LM, Kelman Z; Kelman (2004). "Multiple origins of replication in archaea". Trends Microbiol. 12 (9): 399–401. doi:10.1016/j.tim.2004.07.001. PMID 15337158.
- ^ Lindås AC, Karlsson EA, Lindgren MT, Ettema TJ, Bernander R. (2008). "A unique cell division machinery in the Archaea". Proc Natl Acad Sci U S A. 105 (48): 18942–6. Bibcode:2008PNAS..10518942L. doi:10.1073/pnas.0809467105. PMID 18987308.
- ^ Samson RY, Obita T, Freund SM, Williams RL, Bell SD. (2008). "A role for the ESCRT system in cell division in archaea". Science. 12 (322): 5908. Bibcode:2008Sci...322.1710S. doi:10.1126/science.1165322. PMID 19008417.
- ^ Pelve EA, Lindås AC, Martens-Habbena W, de la Torre JR, Stahl DA, Bernander R. (2011). "Cdv-based cell division and cell cycle organization in the thaumarchaeon Nitrosopumilus maritimus". Mol Microbiol. 82 (3): 555–566. doi:10.1111/j.1365-2958.2011.07834.x. PMID 21923770.
- ^ Caspi, Yaron; Dekker, Cees (2018-03-02). "Dividing the Archaeal Way: The Ancient Cdv Cell-Division Machinery". Frontiers in Microbiology. 9: 174. doi:10.3389/fmicb.2018.00174. ISSN 1664-302X. PMC 5840170 . PMID 29551994.
- ^ Onyenwoke RU, Brill JA, Farahi K, Wiegel J; Brill; Farahi; Wiegel (2004). "Sporulation genes in members of the low G+C Gram-type-positive phylogenetic branch ( Firmicutes)". Arch. Microbiol. 182 (2–3): 182–92. doi:10.1007/s00203-004-0696-y. PMID 15340788.
- ^ Kostrikina NA, Zvyagintseva IS, Duda VI.; Zvyagintseva; Duda (1991). "Cytological peculiarities of some extremely halophilic soil archaeobacteria". Arch. Microbiol. 156 (5): 344–49. doi:10.1007/BF00248708.
- ^ DeLong EF, Pace NR; Pace (2001). "Environmental diversity of bacteria and archaea". Syst. Biol. 50 (4): 470–8. doi:10.1080/106351501750435040. PMID 12116647.
- ^ a b Pikuta EV, Hoover RB, Tang J; Hoover; Tang (2007). "Microbial extremophiles at the limits of life". Crit. Rev. Microbiol. 33 (3): 183–209. doi:10.1080/10408410701451948. PMID 17653987.
- ^ Adam, Panagiotis S; Borrel, Guillaume; Brochier-Armanet, Céline; Gribaldo, Simonetta (2017-11). "The growing tree of Archaea: new perspectives on their diversity, evolution and ecology". The ISME Journal (dalam bahasa Inggris). 11 (11): 2407–2425. doi:10.1038/ismej.2017.122. ISSN 1751-7362. PMC 5649171 . PMID 28777382.
- ^ Madigan MT, Martino JM (2006). Brock Biology of Microorganisms (edisi ke-11th). Pearson. hlm. 136. ISBN 0-13-196893-9.
- ^ Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K; Nakamura; Toki; Tsunogai; Miyazaki; Miyazaki; Hirayama; Nakagawa; Nunoura; Horikoshi (2008). "Cell proliferation at 122 °C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation". Proceedings of the National Academy of Sciences of the United States of America. 105 (31): 10949–54. Bibcode:2008PNAS..10510949T. doi:10.1073/pnas.0712334105. PMC 2490668 . PMID 18664583.
- ^ Ciaramella M, Napoli A, Rossi M; Napoli; Rossi (February 2005). "Another extreme genome: how to live at pH 0". Trends Microbiol. 13 (2): 49–51. doi:10.1016/j.tim.2004.12.001. PMID 15680761.
- ^ Javaux EJ (2006). "Extreme life on Earth—past, present and possibly beyond". Res. Microbiol. 157 (1): 37–48. doi:10.1016/j.resmic.2005.07.008. PMID 16376523.
- ^ Nealson KH (January 1999). "Post-Viking microbiology: new approaches, new data, new insights" (PDF). Origins of Life and Evolution of Biospheres. 29 (1): 73–93. doi:10.1023/A:1006515817767. PMID 11536899.
- ^ Davies PC (1996). "The transfer of viable microorganisms between planets". Ciba Found. Symp. 202: 304–14; discussion 314–7. PMID 9243022.
- ^ López-García P, López-López A, Moreira D, Rodríguez-Valera F; López-López; Moreira; Rodríguez-Valera (July 2001). "Diversity of free-living prokaryotes from a deep-sea site at the Antarctic Polar Front". FEMS Microbiol. Ecol. 36 (2–3): 193–202. doi:10.1016/s0168-6496(01)00133-7. PMID 11451524.
- ^ Karner MB, DeLong EF, Karl DM; Delong; Karl (2001). "Archaeal dominance in the mesopelagic zone of the Pacific Ocean". Nature. 409 (6819): 507–10. Bibcode:2001Natur.409..507K. doi:10.1038/35054051. PMID 11206545.
- ^ Giovannoni SJ, Stingl U.; Stingl (2005). "Molecular diversity and ecology of microbial plankton". Nature. 427 (7057): 343–8. Bibcode:2005Natur.437..343G. doi:10.1038/nature04158. PMID 16163344.
- ^ DeLong EF, Karl DM; Karl (September 2005). "Genomic perspectives in microbial oceanography". Nature. 437 (7057): 336–42. Bibcode:2005Natur.437..336D. doi:10.1038/nature04157. PMID 16163343.
- ^ Agogué, H; Brink, M; Dinasquet, J; Herndl, GJ (2008). "Major gradients in putatively nitrifying and non-nitrifying Archaea in the deep North Atlantic". Nature. 456 (7223): 788–791. Bibcode:2008Natur.456..788A. doi:10.1038/nature07535. PMID 19037244.
- ^ Teske A, Sørensen KB; Sørensen (January 2008). "Uncultured archaea in deep marine subsurface sediments: have we caught them all?". ISME J. 2 (1): 3–18. doi:10.1038/ismej.2007.90. PMID 18180743.
- ^ Lipp JS, Morono Y, Inagaki F, Hinrichs KU; Morono; Inagaki; Hinrichs (July 2008). "Significant contribution of Archaea to extant biomass in marine subsurface sediments". Nature. 454 (7207): 991–4. Bibcode:2008Natur.454..991L. doi:10.1038/nature07174. PMID 18641632.
- ^ Danovaro, Roberto; Dell’Anno, Antonio; Corinaldesi, Cinzia; Rastelli, Eugenio; Cavicchioli, Ricardo; Krupovic, Mart; Noble, Rachel T.; Nunoura, Takuro; Prangishvili, David (2016-10). "Virus-mediated archaeal hecatomb in the deep seafloor". Science Advances (dalam bahasa Inggris). 2 (10): e1600492. doi:10.1126/sciadv.1600492. ISSN 2375-2548. PMC 5061471 . PMID 27757416.
- ^ Liu, Xiaobo; Pan, Jie; Liu, Yang; Li, Meng; Gu, Ji-Dong (2018-10). "Diversity and distribution of Archaea in global estuarine ecosystems". Science of The Total Environment (dalam bahasa Inggris). 637-638: 349–358. doi:10.1016/j.scitotenv.2018.05.016.
- ^ Cabello P, Roldán MD, Moreno-Vivián C; Roldán; Moreno-Vivián (November 2004). "Nitrate reduction and the nitrogen cycle in archaea". Microbiology (Reading, Engl.). 150 (Pt 11): 3527–46. doi:10.1099/mic.0.27303-0. PMID 15528644.
- ^ Mehta MP, Baross JA; Baross (December 2006). "Nitrogen fixation at 92 degrees C by a hydrothermal vent archaeon". Science. 314 (5806): 1783–6. Bibcode:2006Sci...314.1783M. doi:10.1126/science.1134772. PMID 17170307.
- ^ Coolen MJ, Abbas B, van Bleijswijk J; et al. (April 2007). "Putative ammonia-oxidizing Crenarchaeota in suboxic waters of the Black Sea: a basin-wide ecological study using 16S ribosomal and functional genes and membrane lipids". Environ. Microbiol. 9 (4): 1001–16. doi:10.1111/j.1462-2920.2006.01227.x. PMID 17359272.
- ^ Leininger S, Urich T, Schloter M; et al. (August 2006). "Archaea predominate among ammonia-oxidizing prokaryotes in soils". Nature. 442 (7104): 806–9. Bibcode:2006Natur.442..806L. doi:10.1038/nature04983. PMID 16915287.
- ^ Baker, B. J; Banfield, J. F (2003). "Microbial communities in acid mine drainage". FEMS Microbiology Ecology. 44 (2): 139–152. doi:10.1016/S0168-6496(03)00028-X. PMID 19719632.
- ^ Schimel J (August 2004). "Playing scales in the methane cycle: from microbial ecology to the globe". Proceedings of the National Academy of Sciences of the United States of America. 101 (34): 12400–1. Bibcode:2004PNAS..10112400S. doi:10.1073/pnas.0405075101. PMC 515073 . PMID 15314221.
- ^ Eckburg P, Lepp P, Relman D; Lepp; Relman (2003). "Archaea and their potential role in human disease". Infect Immun. 71 (2): 591–6. doi:10.1128/IAI.71.2.591-596.2003. PMC 145348 . PMID 12540534.
- ^ Cavicchioli R, Curmi P, Saunders N, Thomas T; Curmi; Saunders; Thomas (2003). "Pathogenic archaea: do they exist?". BioEssays. 25 (11): 1119–28. doi:10.1002/bies.10354. PMID 14579252.
- ^ Lepp P, Brinig M, Ouverney C, Palm K, Armitage G, Relman D; Brinig; Ouverney; Palm; Armitage; Relman (2004). "Methanogenic Archaea and human periodontal disease". Proceedings of the National Academy of Sciences of the United States of America. 101 (16): 6176–81. Bibcode:2004PNAS..101.6176L. doi:10.1073/pnas.0308766101. PMC 395942 . PMID 15067114.
- ^ Vianna ME, Conrads G, Gomes BP, Horz HP; Conrads; Gomes; Horz (April 2006). "Identification and quantification of archaea involved in primary endodontic infections". J. Clin. Microbiol. 44 (4): 1274–82. doi:10.1128/JCM.44.4.1274-1282.2006. PMC 1448633 . PMID 16597851.
- ^ Jahn U, Gallenberger M, Paper W; et al. (March 2008). "Nanoarchaeum equitans and Ignicoccus hospitalis: new insights into a unique, intimate association of two archaea". J. Bacteriol. 190 (5): 1743–50. doi:10.1128/JB.01731-07. PMC 2258681 . PMID 18165302.
- ^ Moissl-Eichinger C, Pausan M, Taffner J, Berg G, Bang C, Schmitz RA (Januari 2018). "Archaea Are Interactive Components of Complex Microbiomes". Trends in Microbiology. 26 (1): 70–85. doi:10.1016/j.tim.2017.07.004. PMID 28826642.
- ^ Chaban B, Ng SY, Jarrell KF; Ng; Jarrell (February 2006). "Archaeal habitats—from the extreme to the ordinary". Can. J. Microbiol. 52 (2): 73–116. doi:10.1139/w05-147. PMID 16541146.
- ^ Schink B (June 1997). "Energetics of syntrophic cooperation in methanogenic degradation". Microbiol. Mol. Biol. Rev. 61 (2): 262–80. PMC 232610 . PMID 9184013.
- ^ Lange, M; Westermann, P; Ahring, BK (2005). "Archaea in protozoa and metazoa". Applied Microbiology and Biotechnology. 66 (5): 465–474. doi:10.1007/s00253-004-1790-4. PMID 15630514.
- ^ van Hoek AH, van Alen TA, Sprakel VS; et al. (1 February 2000). "Multiple acquisition of methanogenic archaeal symbionts by anaerobic ciliates". Mol. Biol. Evol. 17 (2): 251–8. doi:10.1093/oxfordjournals.molbev.a026304. PMID 10677847.
- ^ Preston, C.M; Wu, K.Y; Molinski, T.F; Delong, E.F (1996). "A psychrophilic crenarchaeon inhabits a marine sponge: Cenarchaeum symbiosum gen. nov., sp. nov". Proceedings of the National Academy of Sciences of the United States of America. 93 (13): 6241–6. Bibcode:1996PNAS...93.6241P. doi:10.1073/pnas.93.13.6241. PMC 39006 . PMID 8692799.
- ^ Eckburg PB, Bik EM, Bernstein CN; et al. (June 2005). "Diversity of the human intestinal microbial flora". Science. 308 (5728): 1635–8. Bibcode:2005Sci...308.1635E. doi:10.1126/science.1110591. PMC 1395357 . PMID 15831718.
- ^ Samuel BS, Gordon JI; Gordon (June 2006). "A humanized gnotobiotic mouse model of host-archaeal-bacterial mutualism". Proceedings of the National Academy of Sciences of the United States of America. 103 (26): 10011–6. Bibcode:2006PNAS..10310011S. doi:10.1073/pnas.0602187103. PMC 1479766 . PMID 16782812.
- ^ Bang C, Schmitz RA (2015). "Archaea associated with human surfaces: not to be underestimated". FEMS Microbiology Reviews. doi:10.1093/femsre/fuv010. PMID 25907112.
- ^ Wegley, L; Yu, Y; Breitbart, M; Casas, V; Kline, D.I; Rohwer, F (2004). "Coral-associated Archaea" (PDF). Marine Ecology Progress Series. 273: 89–96. doi:10.3354/meps273089. Diarsipkan dari versi asli (PDF) tanggal 2008-09-11. Diakses tanggal 2015-12-17.
- ^ Chelius MK, Triplett EW (April 2001). "The Diversity of Archaea and Bacteria in Association with the Roots of Zea mays L". Microb. Ecol. 41 (3): 252–63. doi:10.1007/s002480000087. JSTOR 4251818. PMID 11391463.
- ^ Simon HM, Dodsworth JA, Goodman RM; Dodsworth; Goodman (October 2000). "Crenarchaeota colonize terrestrial plant roots". Environ. Microbiol. 2 (5): 495–505. doi:10.1046/j.1462-2920.2000.00131.x. PMID 11233158.
- ^ Breithaupt H (2001). "The hunt for living gold. The search for organisms in extreme environments yields useful enzymes for industry". EMBO Rep. 2 (11): 968–71. doi:10.1093/embo-reports/kve238. PMC 1084137 . PMID 11713183.
- ^ a b Egorova K, Antranikian G; Antranikian (2005). "Industrial relevance of thermophilic Archaea". Current Opinion in Microbiology. 8 (6): 649–55. doi:10.1016/j.mib.2005.10.015. PMID 16257257.
- ^ Synowiecki J, Grzybowska B, Zdziebło A; Grzybowska; Zdziebło (2006). "Sources, properties and suitability of new thermostable enzymes in food processing". Crit Rev Food Sci Nutr. 46 (3): 197–205. doi:10.1080/10408690590957296. PMID 16527752.
- ^ Jenney FE, Adams MW; Adams (January 2008). "The impact of extremophiles on structural genomics (and vice versa)". Extremophiles. 12 (1): 39–50. doi:10.1007/s00792-007-0087-9. PMID 17563834.
- ^ Schiraldi C, Giuliano M, De Rosa M; Giuliano; De Rosa (2002). "Perspectives on biotechnological applications of archaea" (PDF). Archaea. 1 (2): 75–86. doi:10.1155/2002/436561. PMC 2685559 . PMID 15803645. Diarsipkan dari versi asli (PDF) tanggal 2013-08-26. Diakses tanggal 2015-12-17.
- ^ Norris PR, Burton NP, Foulis NA; Burton; Foulis (2000). "Acidophiles in bioreactor mineral processing". Extremophiles. 4 (2): 71–6. doi:10.1007/s007920050139. PMID 10805560.
- ^ Shand RF; Leyva KJ (2008). "Archaeal Antimicrobials: An Undiscovered Country". Dalam Blum P (ed.). Archaea: New Models for Prokaryotic Biology. Caister Academic Press. ISBN 978-1-904455-27-1.
Bacaan lebih lanjut
- Howland, John L. (2000). The Surprising Archaea: Discovering Another Domain of Life. Oxford University. ISBN 978-0-19-511183-5.
- Martinko JM, Madigan MT (2005). Brock Biology of Microorganisms (edisi ke-11th). Englewood Cliffs, N.J: Prentice Hall. ISBN 0-13-144329-1.
- Garrett RA, Klenk H (2005). Archaea: Evolution, Physiology and Molecular Biology. WileyBlackwell. ISBN 1-4051-4404-1.
- Cavicchioli R (2007). Archaea: Molecular and Cellular Biology. American Society for Microbiology. ISBN 1-55581-391-7.
- Blum P (editor) (2008). Archaea: New Models for Prokaryotic Biology. Caister Academic Press. ISBN 978-1-904455-27-1.
- Lipps G (2008). "Archaeal Plasmids". Plasmids: Current Research and Future Trends. Caister Academic Press. ISBN 978-1-904455-35-6.
- Sapp, Jan (2009). The New Foundations of Evolution. On the Tree of Life. New York: Oxford University Press. ISBN 0-19-538850-X.
- Schaechter, M (2009). Archaea (Overview) in The Desk Encyclopedia of Microbiology, 2nd edition. San Diego and London: Elsevier Academic Press. ISBN 978-0-12-374980-2.
Pranala luar
| Cari tahu mengenai Arkea pada proyek-proyek Wikimedia lainnya: | |
| Definisi dan terjemahan dari Wiktionary | |
| Gambar dan media dari Commons | |
| Berita dari Wikinews | |
| Kutipan dari Wikiquote | |
| Teks sumber dari Wikisource | |
| Buku dari Wikibuku | |
Umum
- Introduction to the Archaea, ecology, systematics and morphology
- Oceans of Archaea[pranala nonaktif permanen] – E.F. DeLong, ASM News, 2003
Klasifikasi
- NCBI taxonomy page on Archaea
- Genera of the domain Archaea – list of Prokaryotic names with Standing in Nomenclature
- Shotgun sequencing finds nanoorganisms – discovery of the ARMAN group of archaea
Genomika
- Browse any completed archaeal genome at UCSC
- Comparative Analysis of Archaeal Genomes (at DOE's IMG system)